

2012.12.23
PowerToolは2017.5サポートが終了しました。機能がver.5に統合されたのがその理由です。今後のバージョンアップ、サポートはありませんのでご了承ください。
HSPiPというのは、ハンセンの溶解度パラメータ(HSP)を実践的(in Practice) に扱うパッケージソフトです。 HSPiPとはどんなソフトウエアーなのか、知りたい方はこちらのページを参照してください。
HSPiP ver. 4.0に付属するしていたパワーツールは次の4つです。
SphereViewer(hsdファイルのビュアー)
Draw to Smiles (SMILES構造式を得る為のGUIプログラム)
Y-Predict(YMB、物性推算プログラムのGUIプログラム)
Y-Fit(ハンセンの溶解球を探索するプログラム)
このパワーツールはHTML5/JavaScriptで開発されています。
SphereViewerはPirikaのHPでも多くのデモがあるので、おなじみでしょう。
3次元のハンセン空間中に溶媒、溶質がちりばめられ、マウス操作で回転させたり、マウスで溶媒をクリックすると溶媒名が表示されたりします。
このHPで使っているSphereViewerは溶媒や溶質のデータもJavaScriptのプログラムとして持たせてあるので、表示効果まで設定した上で配布できるので便利です。
しかし、溶媒や溶質のデータをJavaScriptのプログラムとした時に(ここまでは自動で行う)、それをコピーしてファイル名をつけて所定の場所にセーブしなければなりません。
それを行うのはユーザーには無理だろうという先生の意見で、今回提供するのは単なるビュアーです。
ビュアー自体は、コピーを作り配布するのは自由です。ビュアーにhsdデータをつけて送れば、HSPiPをお持ちでない方もハンセン空間での溶媒や溶質の振る舞いを眺める事ができます。Helpファイルはこちらを参照してください。
さらに高度な使い方をしたい(ファイルのセーブや配置は自分でできる)、というフィードバックが多ければY-Viewer+の形で提供しようかと思います。
以下のプログラムは、HSPiPを購入した本人のみが利用できるパワーツールです。使い始めるのにHSPiPのライセンスファイルが必要になります。
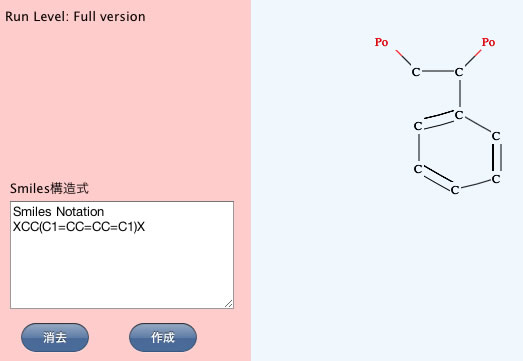
HSPiPでは、分子構造の入力はSmilesの構造式、InCheの構造式、MOLファイルからの入力のみで、GUIでの入力には対応していません。
しかしポリマーのSMILESを入力する場合にはダミー原子(X)を式の両端に配置しなくてはならず、それを手作業で行うのは非常に困難です。
このパワーツールは、キャンバスに描いた分子構造をSMILESの構造式に変換します。また、その構造をデータベースから探索し、HCodeやPCodeがあればそれを返します。2次元の描画ソフトの基本となります。Helpファイルはこちらを参照してください。
HCodeというのはHansen Codeの省略形で、各化合物について定められています。
最初、HSPiPになれないうちは、化合物リスト(大きい方は10000以上)の中から化合物を見つけるのが大変でしょう。
化合物の名称、CAS番号で探すにしても、複数の呼び名、CAS番号があり、なかなかうまく見つからない事が多いと思います。
しかし、例えば化合物のHSP値が変わったり、拡張された場合に、変更履歴はHCodeベースで行われるので、番号を適当につけるのは好ましくありません。
そこで、描かれた分子の情報から化合物リストをサーチして候補を探しだす機能を作成しました。
異性体など複数の候補が出てきてしまいますが、小-中規模分子なら容易に探すことができるでしょう。
Draw2Smilesに搭載されました。
PCodeというのは、ポリマー用のコードです。
ポリマーの場合、輪をかけて名称などで探すのは大変でしょう。
ポリマーを描画して繰り返し末端に印をつけるだけでポリマーのPCodeを探索してくれるので非常に便利に使えます。
Draw2Smilesに搭載されました。
HSPiPに搭載されているYMBはGUIを用いた物性推算には対応していません。
しかし、将来的には物性推算は3次元構造に基づいて行いたいので、YMBをHSPiP本体から切り離し、Y-Predict、独立のモジュールとして開発する事になりました。
また、バージョン間の違いを保持するため、バージョンをプルダウンメニューで選択できるようにしてあります。
また、2次元QEQ法を用いて原子の電荷を計算しています。この計算には分子に水素を付加する必要がありますが、プログラム中で自動的に水素を付加しています(表示はされません)。
電荷が必要ない場合にはチェックボックスを外してください。
使い方はこちらを参照してください。企業からの訪問者でPirikaの物性推算式を使いたい場合にはこれをお使いください。
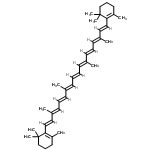
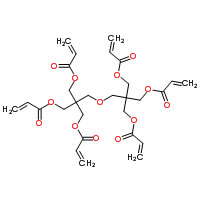
HSPをSmilesの構造式から推算するY-MBの機能で、ある特殊な構造(ビタミン類縁体など)を計算すると計算結果がおかしくなるとの指摘があった。(2重結合の数が多い以下のようなもので結果がおかしくなる) それを計算するver. 3.3 はY-Predict に搭載されました。
ハンセンの溶解球を探索するプログラムはHSPiPの中でも中核のプログラムです。遺伝的アルゴリズム(GA)を使ったアルゴリズムは、JAVAで開発したものをC#に書き換えて先生の所に送ってあるのですけど、何故だか知らないが非常に動作が遅くなります。JavaScript版のプログラムと比べても非常に遅いので、探索の深さを限っています。しかし、だんだん高度な探索が必要になり、この遅さは致命的ですのでC#をあきらめ、HSPiPとは別のパワーツールとして開発する事になりました。 使い方はこちらを参照してください。
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください) メールの件名は[pirika]で始めてください。