2025.5.1
pirika.comで化学
>チャピエモン-3rd Pirika Origin (C-3PO)
> ハンセン溶解度パラメータ (HSP)
>HSPiP(実践ハンセン溶解度パラメータ)ソフトウエアー
> HSPiPの購入方法
> HSPiPを用いた解析例
>基礎 >応用 >ポリマー >医薬品など >環境・溶媒設計 >分析
>その他 >DIY/YMB >アバターチュートリアル >次世代に向けて
>次世代HSP2技術
> 化学全般
>Pirika Pro ツール群
ブログ
業務案内
お問い合わせ
注意:HSPiPに搭載の機能ではありません
JOURNAL OF MACROMOLECULAR SCIENCE Part A—Pure and Applied Chemistry Vol. A41, No. 8, pp. 971–979, 2004
Solubility Parameters of Cross-Linked Poly(N-Vinyl-2-pyrrolidone-co-crotonic Acid) Copolymers Prepared by g-Ray-Induced Polymerization Technique
Tuncer C ̧ aykara
この論文から、Poly(N-Vinyl-2-pyrrolidone-co-crotonic Acid)の膨潤性のデータを入手した。
この論文自体は、ポリビニルピロリドンとハンセン溶解度パラメータ(HSP)で2011年に解析している。
今回は次世代 HSP2を使って解析し直してみた。
PVP:Poly(N-Vinyl-2-pyrrolidone)は親水性の高いポリマーで、生体親和性が高い。医薬品のキャリアー、酵素修飾、生体用の接着剤などに使われる。
ハイドロゲルとして使う場合には、吸水性を高めるためにカルボン酸基を含むモノマーと共重合することが多い。
クロトン酸量と膨潤性
Pirika25Pro4MIのユーザーは次のデータをエクセルにコピペして、説明通りにやってみて欲しい。
クロトン酸の導入効果
先のテーブルをグラフ化すると次のようになる。
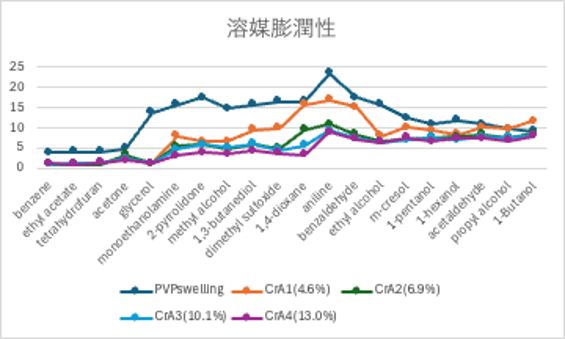
ほとんどの溶媒で、クロトン酸を増やすにつれ膨潤性が低下している。これは何故だろう?
ハンセンの溶解度パラメータで説明できるだろうか?
33式を計算する用のNew Sphereフォーマット
解析用のデータを作り、33式を計算する方法はV-tubeの動画を作ってある。
33式の詳しい説明は「HSP距離の33式詳説」を参照して欲しい。
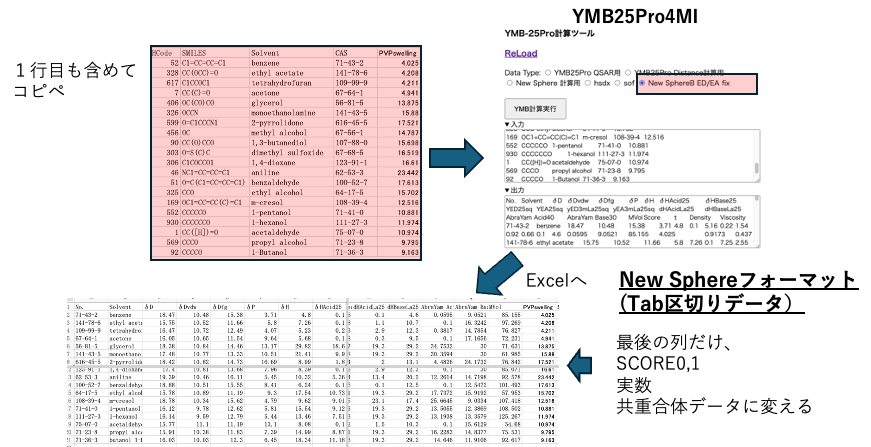
HSPiP用のデータはxmlフォーマットで表計算ソフトとの相性は悪い。
New Sphereフォーマットはタブ区切りの表形式のデータなので、Scoreの入れ替えはとても楽だ。
溶媒を3次元空間にプロットしてみよう
PVPのホモポリマーやクロトン酸との共重合ポリマーの膨潤性を扱いたい訳だが、用いる溶媒は同じだ。これらの溶媒を3次元のハンセン空間にプロットしてみよう。(ハンセン空間は狭義にはdD, dP, dHを軸にした空間を指すが、ここでは拡張HSPもハンセン空間と呼ぶ)Pirika25Pro4MIからSphere Viewer 25を選び上のテキストエリアにNew Sphere フォーマットのデータをペーストしてReadすれば、溶媒が3次元にプロットされる。フォーマットの最後の列がScore 0,1の場合、Score 1のものは青、Score 0のものは赤く表示される。Scoreが実数の場合は、値を6等分して色をつけてある。
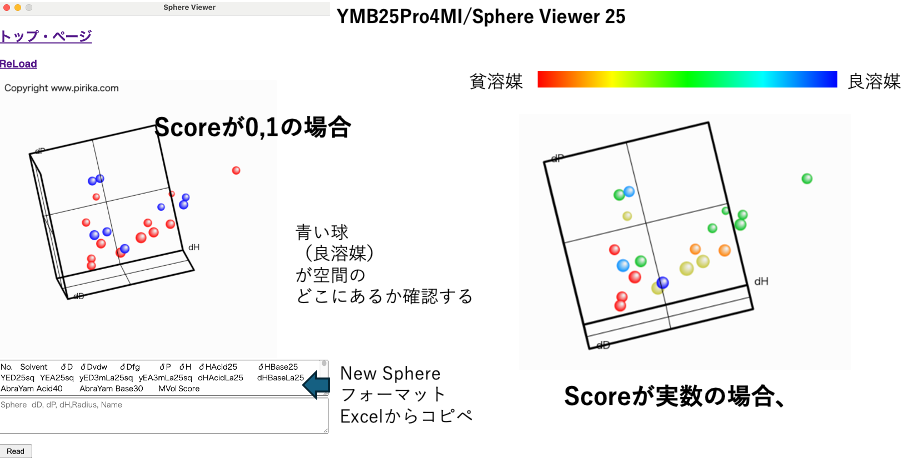
Score 0,1を空間の位置から判断してみよう
膨潤性 赤:9%以下 緑:9%-15% 青:15%以上
小さな球をクリックすると溶媒名が表示される。
Shift Drag:拡大・縮小 Option Drag:移動
一見して、緑と青の球が入り乱れている事がわかるだろう。膨潤性をどこで区切るかは難しい。
溶媒のScoreを良溶媒:1、貧溶媒:0に分ける解析方法は定性的解析と言われる。
例えば、一番シンプルな[dD, dP, dH]で解析すると次のようになる。
PVPホモポリマーの定性的解析
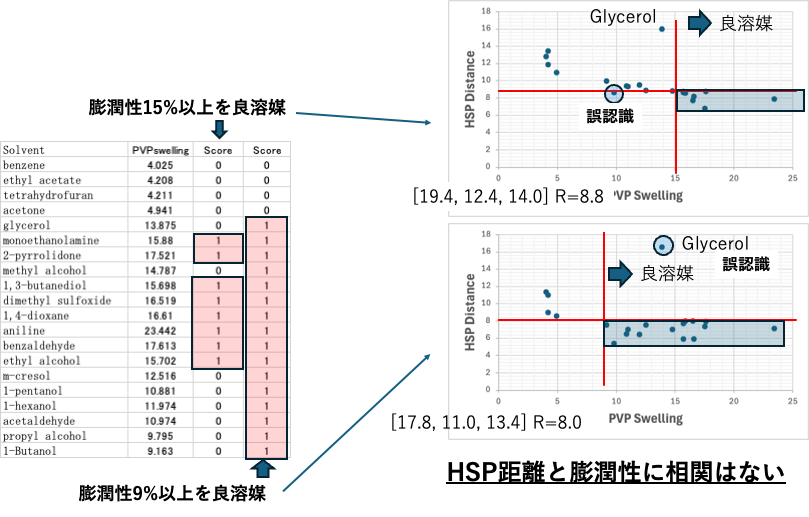
良溶媒の区切りを9%とした時と15%としてハンセンの溶解球の中心を求める。どちらも誤認識(良溶媒なのに球の外に配置、貧溶媒なのに球の内側に配置)は1つなので良いモデルと言える。
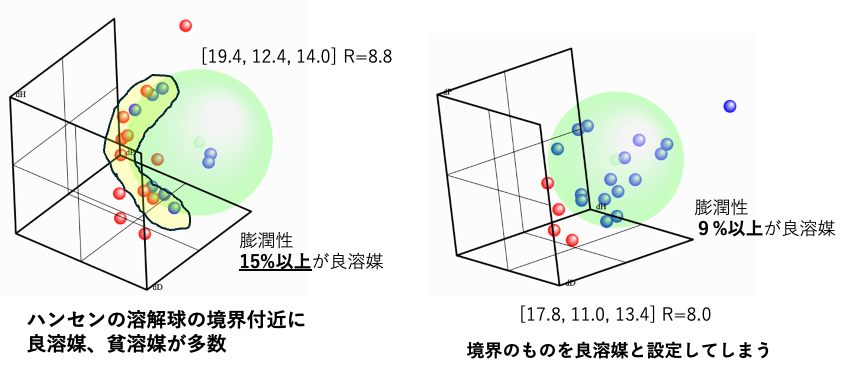
ところが3次元にプロットしてみると意味合いは大きく異なる事がわかる。
膨潤性15%以上が良溶媒とした場合には、9-15%膨潤させる溶媒は溶解球の周辺に張り付くように配置される。それでも誤認識が1つで収まるほど微妙な中心と半径を割り出してしまうのが遺伝的アルゴリズム法(GA法)の凄いところだ。
実際に動かしながら確認してほしい。
15%以上が良溶媒
9%以上が良溶媒
Sphere Viewer 25で溶解球まで描かせたい場合には、下のテキストエリアに
SphereのdD, dP, dH, 半径をタブ区切りで入力する。Sphereは複数入力する事ができる。
PVPホモポリマーの定量的解析
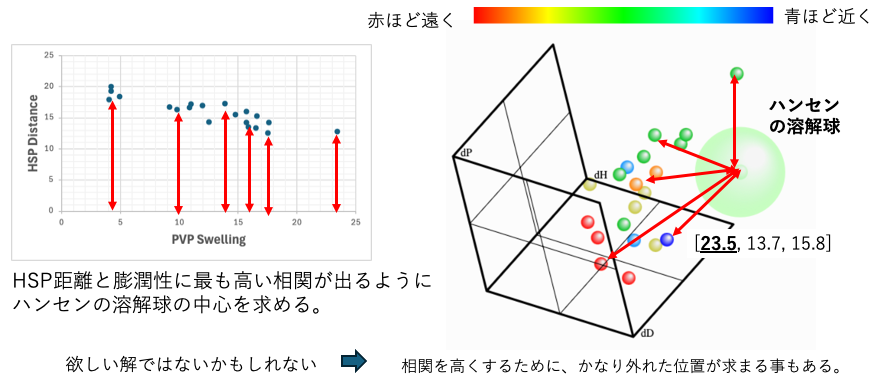
Scoreに実数(良溶媒ほど大きな値になるように調整する必要がある)を入れた場合には、定量的解析を行う。(HSPiPではData Pointsというオプションを使う)
これは、HSP距離と溶解性が一番高い相関(逆相関)となるように溶解球の中心を求める。
実際に動かして確認してみよう。赤などの暖色系統ほど溶解球の中心から遠く、青などの寒色ほど溶解球に近くなるように溶解球の中心を求めている。溶解球の半径は求まらないので、適当な値を入れた。ビジュアル効果を自分で判断して欲しい。
この場合もGA法の探索の凄さなのだが、かなり遠いところに相関係数が高くなる中心を求めることに成功している。
実数Scoreの取り方
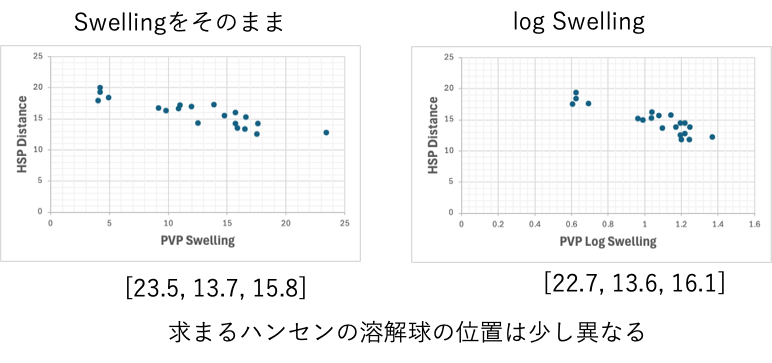
溶解性が一番大きいものと小さいもので100倍変わるようなものは、横軸が離散的になるのでlogを取って圧縮した方が結果は良くなる。この場合logを取ると、より青い球が溶解球のそばに集まる事がわかる。
定性的、定量的の違い
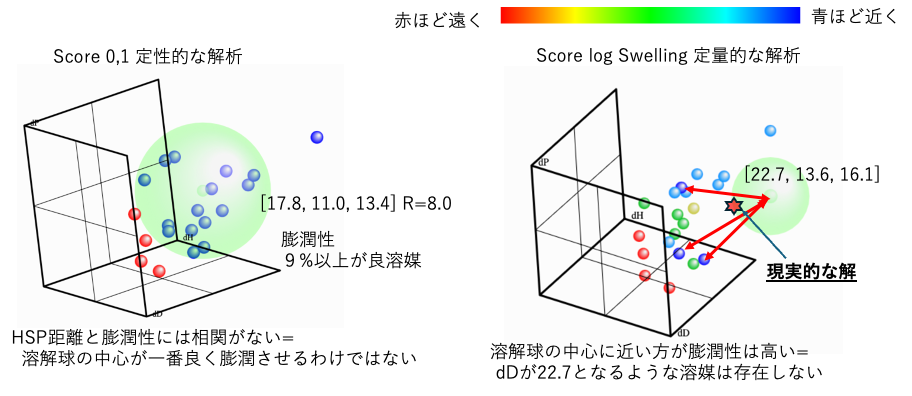
定性的解析では、一番よく溶かす溶媒は設計できない。
定量的解析では、今回の場合、dDが20を超えるものを示唆しているが、そのような溶媒はほとんど存在しない。現実的な解は中心からズレた位置(相関係数は悪い位置)になるのだろう。
溶解球の中心が実在の溶媒から離れている時
そもそも、HSP距離は方向を持たないスカラー量で、それが溶解量と相関があるというのは仮定の話である。離れる方向によって溶解量が変わるなら、高い相関係数を持つものが正しい答えではない。
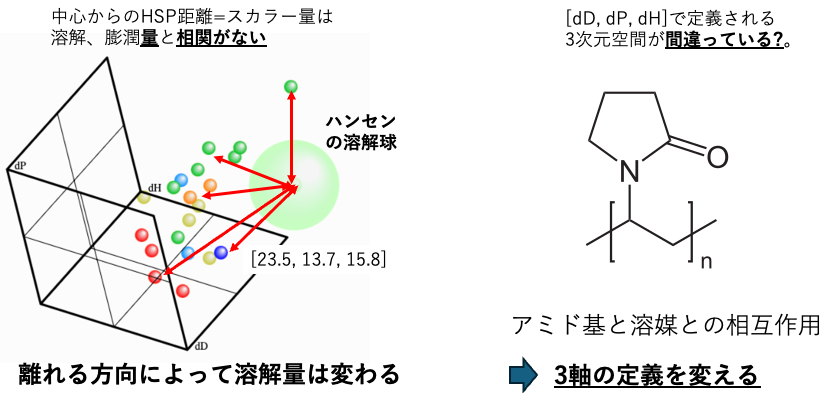
もしくは、[dD, dP, dH]の3次元空間の表記では、この現象を正しく表現できていない可能性もある。
人間は3次元までしか認識できない
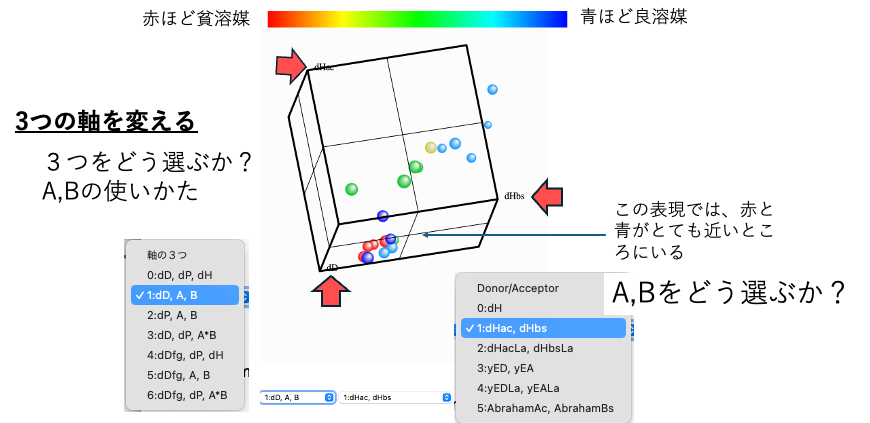
3つの軸に何を置くか、水素結合の分割はどのタイプを選ぶのか、で3次元空間の表現は大きく変わる。
実際に選択して見て欲しい。(この機能はまだPirika25Pro4MIには搭載されていない)
膨潤性が大きい領域での表現力
33式による距離の式を検討すると、33種類の距離の式と膨潤性の関係が得られる。これを図示するにはPirika25Pro4MIの「HSP距離 新33式のグラフ表示」を使う。
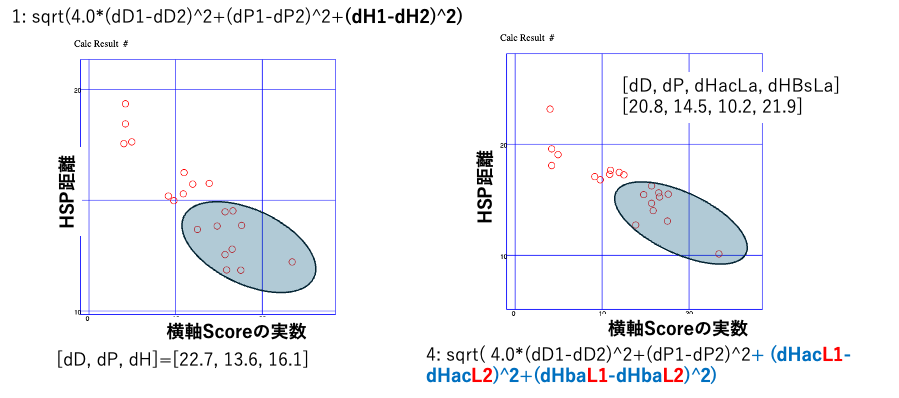
GA法自体は相関係数が大きいものを良い式と判断してしまうが、必ず図示した方が良い。相関係数は大きく外れるものがあると、とても悪くなる。また、用途によっては平均的にバランスの取れた式が良い場合とか、膨潤性が大きいと所のみの表現力が高い方が良い場合もある。自分で見て判断する癖をつけよう。
33式のビュアー使い方
Pirika25Pro4MIには33式のビュアーが搭載されている。特に実数を使った解析を行う場合にはすぐに試してみよう。相関係数だけで判断するのはとても危険だ。

何故表現力が異なるのか3Dで確認
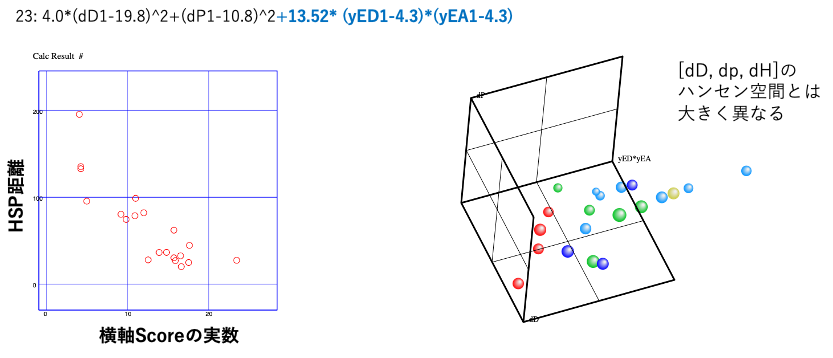
クロトン酸の導入効果
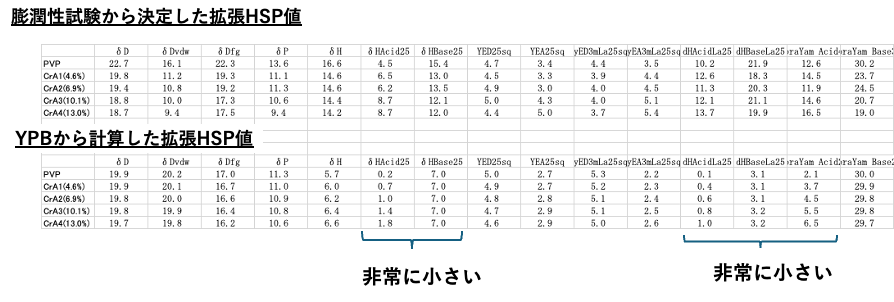
ここまでの話はPVPのホモポリマーの話だ。4種類のCrAとの共重合ポリマーの膨潤性に関しても33式を計算する。計算結果のダイジェストから導入効果を評価する。
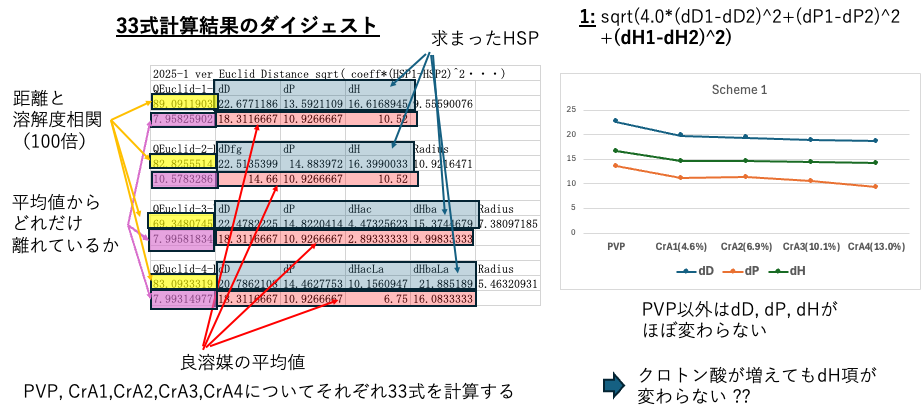
不思議なことに、クラッシクの[dD, dP, dH]はPVP以外はほぼ同じ値になる。クロトン酸が増えてもdH項が変わらないというのは不思議な結果だ。
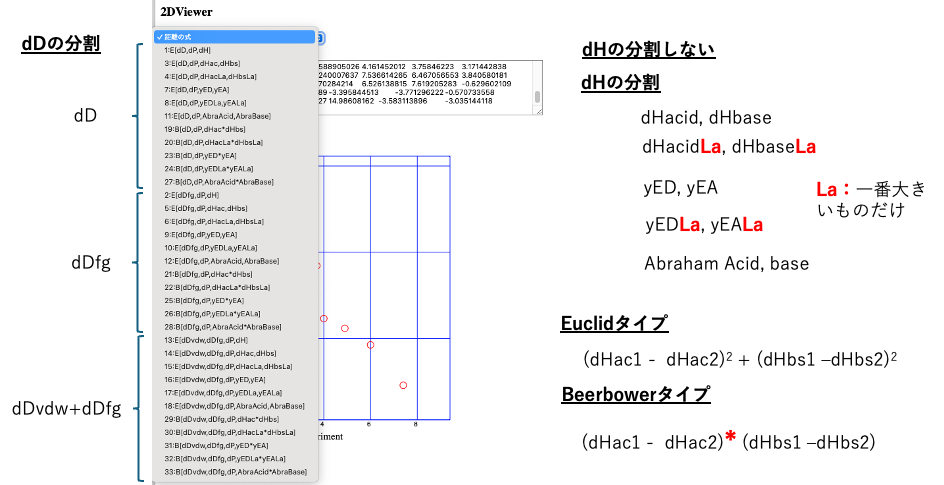
次世代HSP2でdHを分割しなくてはならない
逆に言えば、酸性ポリマーと明らかにわかっているポリマーを旧来の方法で解析しても正しい答えは出ないということだ。
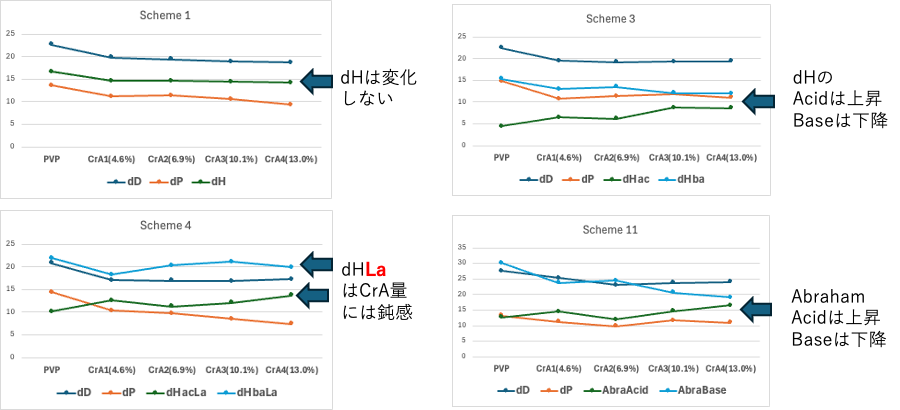
33式の簡単な説明は後につけるが、dHをdHac, dHbsに分けた場合には、クロトン酸が増えるにつれてdHacは大きくなって、dHbsは小さくなる。AbrahamのAcid/Baseに分けた場合も同様だ。溶媒の官能基のうち一番大きな値だけを使うLaにすると、クロトン酸量の違いに対しては鈍感になる。
Euclidタイプを使うか、Beerbowerタイプか?
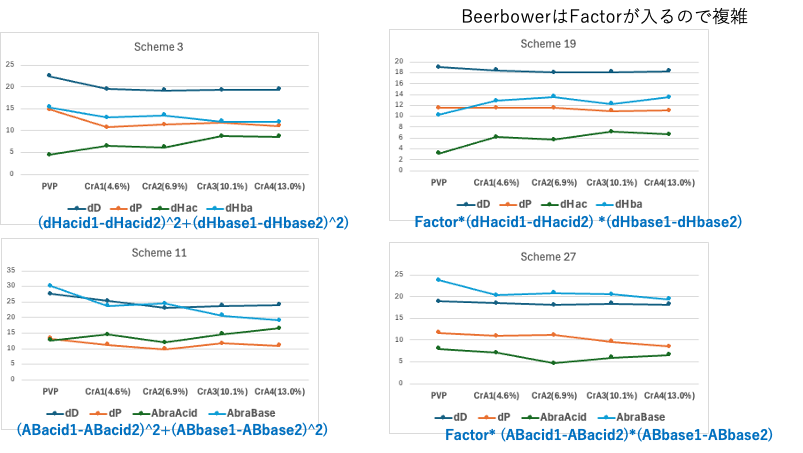
Beerbowerの場合、差分の掛け算に、さらにFactorがかかるために意味合いが複雑になる。この場合にはEuclidタイプでも十分に表現できていると言えそうだ。(金属塩の場合にはEuclidタイプでは表現できないことがある。)
ポリマーの3次元表示
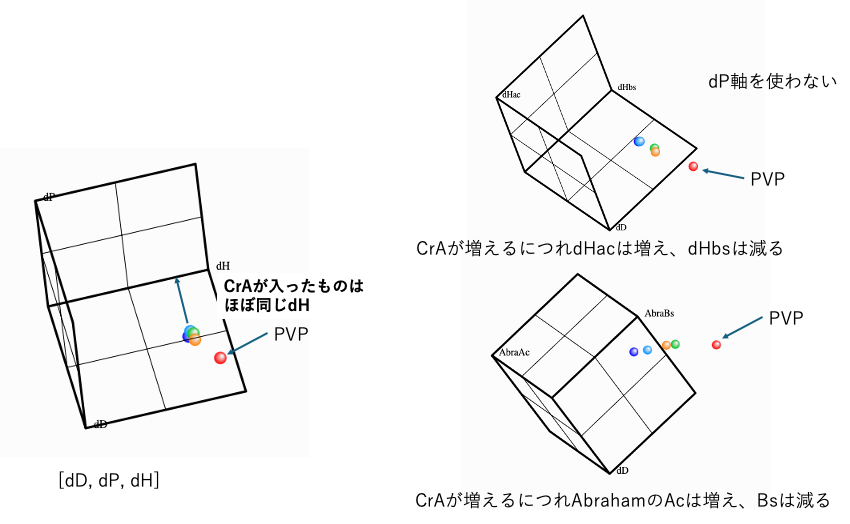
溶媒だけでなくポリマーに関しても3軸を変えながらポリマーを表示させる事ができる。軸の取り方によって表現が大きく変わることが理解できる。
HSPiPではこの細かい解析はできない。(それでも、高いパーフォーマンスを確保できているのでいまだに使われ続けている)
特に特許を書かなくてはならない、もしくは他社のパラメータ特許を回避したい場合には次世代のHSP2が意味を持ってくる。私自身はパラメータ特許は嫌いだ。
パラメータが支持する化合物自体で取ることをお勧めしている。
その時にも、他社が取った範囲外の化合物のdH ac/bsやAbraham のAc/bsからコレコレの化合物が特に良かったと記載した時に(他には理由はオープンにしなくても)大事な範囲の特許が取れると思う。
Y-PBで単独ポリマーの検証
Pirika25Pro4MIにはポリマーのコードを調べる機能が搭載されている。
元々は、私自身がポリマーのDBを作るときに、同じポリマーをどう識別していくか?のために作ったWebアプリだ。ポリマーには多くの異なる名称がある。
ある名称を聞いた時にそれが既にDBにあるかどうかを調べるのはとても大変だ。
それは、複雑な構造式だけがある場合も同様だ。
例えばJSMEで分子を描いただけでDBの登録コードがわかれば、物性値の拡充も圧倒的に楽になる。
Y-PBでポリマーの物性推算式が開発できたのも、先にこのコードを実用化しておいたからだ。
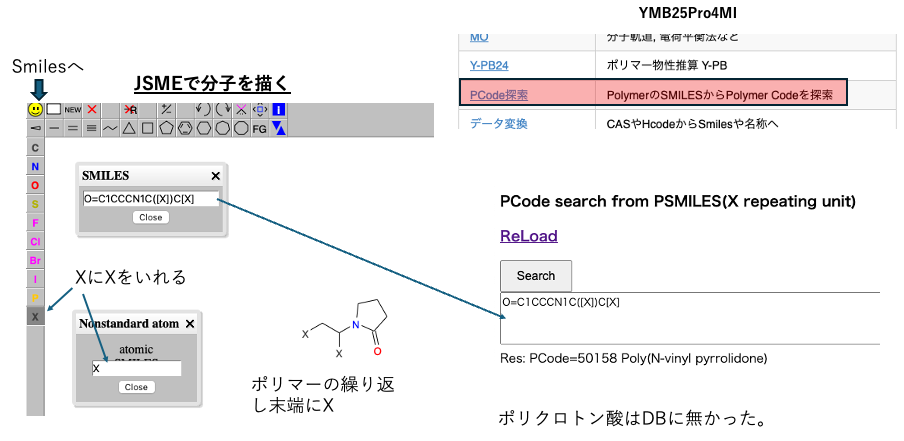
JSME(HSPiPをインストールしていればHSPiP Dataの中に入っているはず)でポリマーの繰り返し末端をXと指定してお絵描きする。得られたポリマーSMILESをPirika25Pro4MIのPCodeサーチにかければ、自分のDBで使っているコードが得られる。
Pcodeを使ってHSPiPのDB検索
得られたPCodeを使ってHSPiPのポリマーDBを検索してみる。
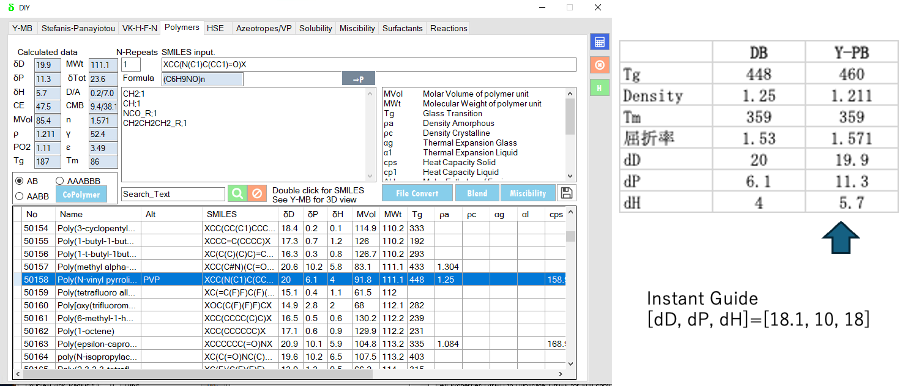
ポリ・クロトン酸は見つからないが、PVPのポリマーはHSPiPに搭載されている。
Y-PBの物性推算値とDB値の違いはすぐに検証する事ができる。
私はHSPiPの開発者でもあるので、Y-PBを開発する用のポリマーDBも収集している。
例えばPVPのホモポリマーのPCodeがわかれば、(存在するなら)溶解性のDBもすぐに見つかる。
pirikaの持つポリマー溶解性DB
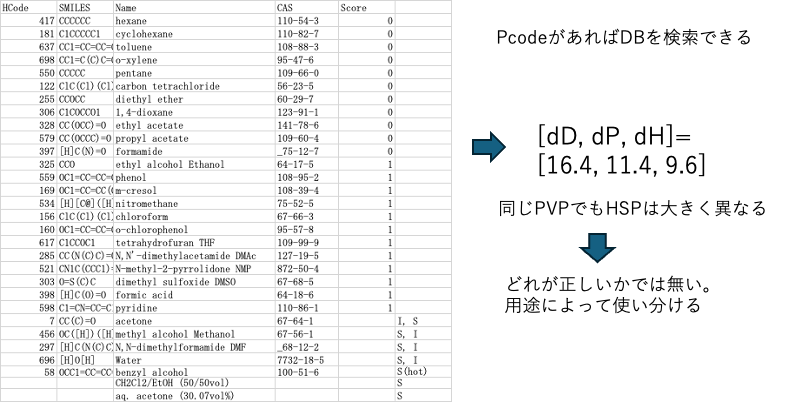
PVPに関してはDBがあった。
このデータが得られればHSPを計算することが可能になる。
(こうしたデータを元にY-PBでHSPを推算式を構築していく)
Y-PB25Proを使った推算値
ポリマーSMILESがあれば、Y-PBプロを使い、ポリマー物性を推算する事ができる。
様々な考え方があり得るが、一つの解釈として、ポリマーの高次構造の影響が考えられる。
ポリマーの立体構造表示
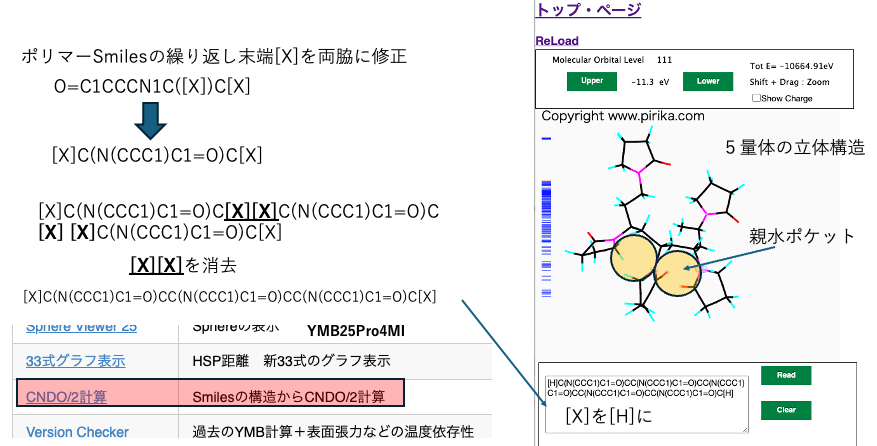
ポリマーSMILESの繰り返し末端を両脇に持ってくる。
手作業では大変なので、Pirika25Pro4MIの5月最新版には変換プログラムを付け加えた。(YPB25Proで普通に物性推算すると変換されたポリマーSMILESも出力される)
その後は例えばポリマーSMILES2つ置き真ん中の[X][X]を消去すれば2量体のポリマーSMILESが得られる。モノマーなどの原料をこの形式で用意しておけば、共重合体や縮合系のポリマーも簡単に作れる。
例えば、5量体のSmilesの構造式を作ってみよう。
[H]C(N(CCC1)C1=O)CC(N(CCC1)C1=O)CC(N(CCC1)C1=O)CC(N(CCC1)C1=O)CC(N(CCC1)C1=O)C[H]
XはHに変えておく。これは3次元構造をRDKitに作らせるためである。
それをPirika25Pro4MIの中にあるCNDO/2計算ソフトに流し込めば3次元構造が得られる。RDKitはMMF94という分子力場法を使って安定構造を計算する。
すると、ピロリドン環は立体障害で少し捻れていく構造が得られる。
そこでアミド基が近くに偏在する親水ポケットができているのでは無いかと想像できる。
YPBではあくまでも官能基でしか判断しない。高次構造が物性に関与するのであれば無力である。クロトン酸の持つカルボキシル基も集まって親水ポケットを作っている可能性が高い。
逆相ミセルを作っているようなイメージだ。
MMF94の計算次第で環が互い違いになったりもするが、親水ポケットは確認できるだろう。
ところで、この論文でγ線を使ったのは何故だろう?
現在、ちまちまとポリマー用のツール、Polymer Studio Proを作成中である。
その中にPOSEIDONというラジカル重合シミュレータがある。(Pirika25Pro4MIには未搭載)
POSEIDONとは POlymer SEquence IDentified ON Mac、でMac上でポリマー中のモノマー・シーケンスを決定する用の自作(pirika.com製)のソフトだ。尾川さんが名付けてくれた。今なら、On the Netか。
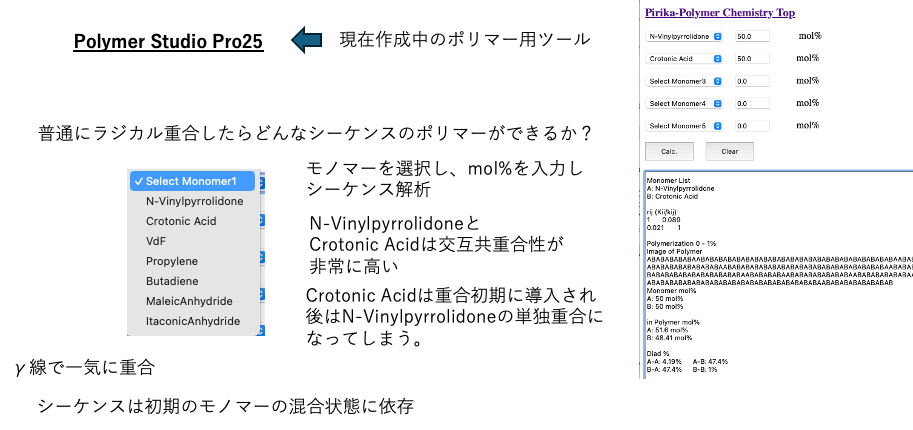
モノマーの量を変えながら重合してみよう。ビニルピロリドンとクロトン酸の比率を幾つにすれば、ポリマーに4%ほどクロトン酸が導入されるだろうか?
この2つのモノマーは非常に交互共重合性が高く、クロトン酸の量を減らしても、非常に早くポリマーに導入されてしまう。残りの重合では、ビニルピロリドンの単独重合になってしまう。
参考までに、POSEIDONではQe Theoryを使ってシーケンスを求めている。

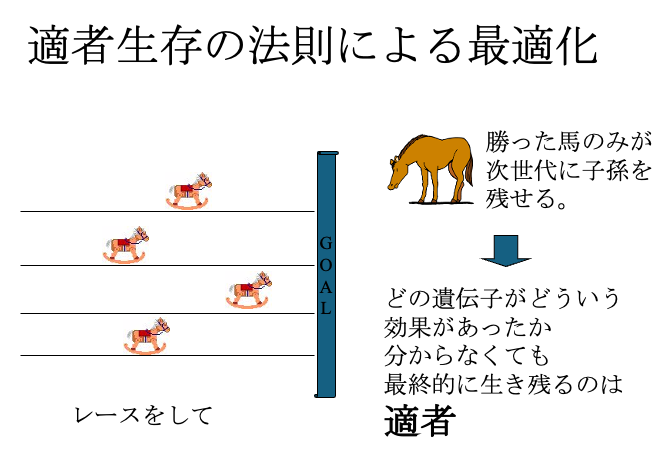
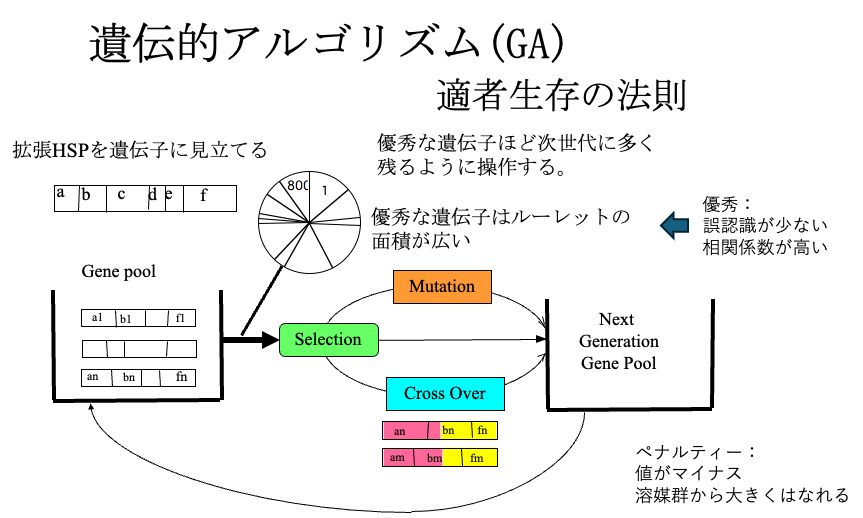
参考:溶解球の探索方法
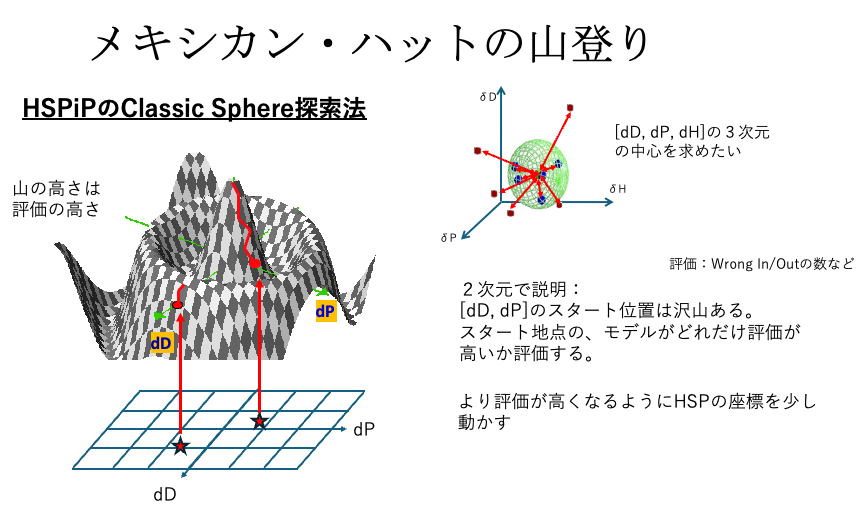
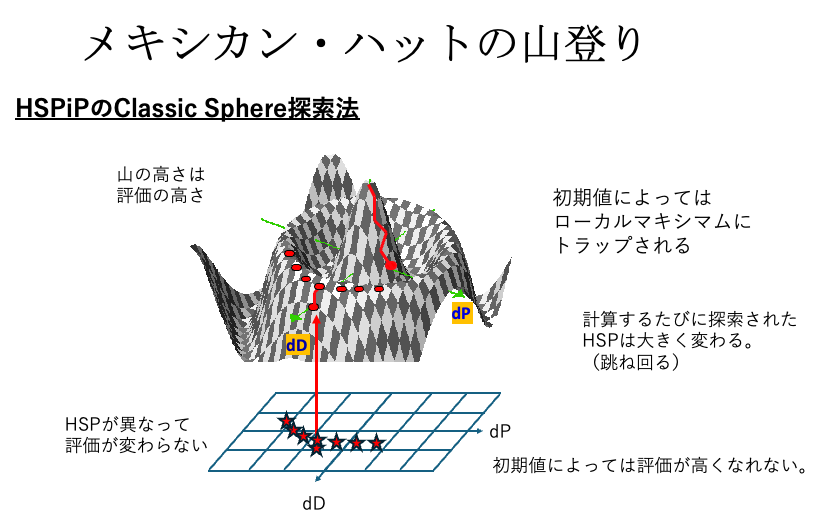
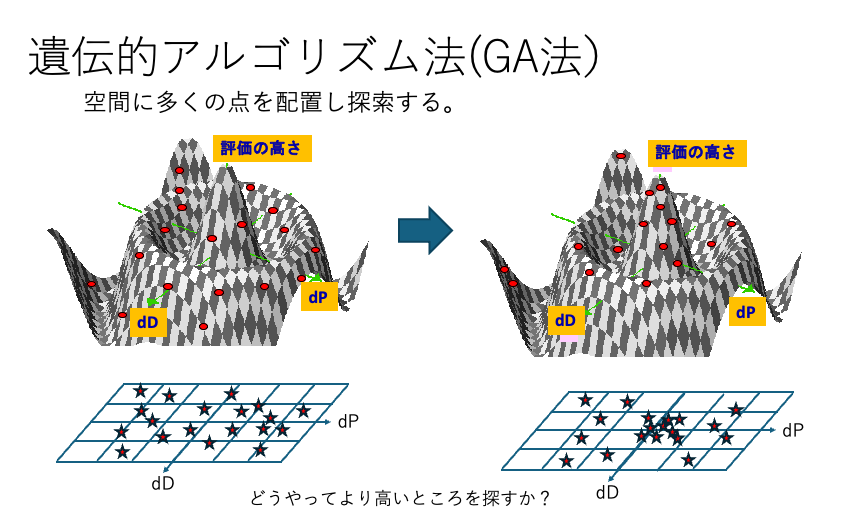
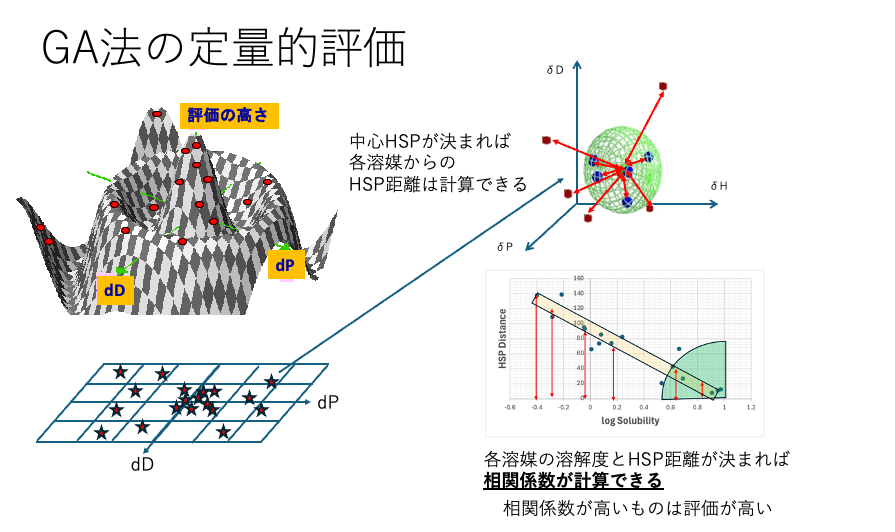
参考:遺伝的アルゴリズム(GA)法
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください
