2022.10.9改訂(2012.4.26)
pirika.comで化学 > 化学全般
> 次世代HSP2, YMB Pro for MI >
> 解析例トップ
DIYトップページ >
医薬品・化粧品トップページ > 経皮吸収型ドラッグデリバリーシステム
TDDS(Transdermal Drug Delivery System)、経皮吸収型ドラッグデリバリーシステムに関する論文を見つけた。Yakugaku Zasshi 127(4) 655-662 (2007) 久光製薬の肥後さんという方の書かれた論文で、TDDSの概略を知るには良い論文だ。
その中に、これまでに開発されてきたTDDSの特徴のまとめがあった。
薬の構造には次のようなものがあるようだ。
次のテーブルの中身をエクセルなどにコピペしておこう。
テーブル
| Hcode | Name | CAS | Smiles | Delivery rate mg/day | MW | MP | logKow |
|---|---|---|---|---|---|---|---|
| 20441 | Clonidine | 4205-90-7 | Clc1c(c(Cl)ccc1)N/C2=N/CCN2 | 0.1 | 230 | 140 | 0.83 |
| 20323 | Estradiol | 50-28-2 | CC12CCC3C(C1CCC2O)CCC4=C3C=CC(=C4)O | 0.05 | 272 | 176 | 2.49 |
| 22349 | Fentanyl | 437-38-7 | O=C(N(c1ccccc1)C3CCN(CCc2ccccc2)CC3)CC | 0.6 | 337 | 83 | 2.93 |
| X1 | Isosorbide dinitrate | 87-33-2 | [O-]N+O[C@H]1[C@H]2OCC@H[C@H]2OC1 | 11 | 236 | 70 | |
| 1198 | Nicotine | 54-11-5 | CN1C@@HCCC1 | 5 | 162 | <-80 | 1.17 |
| 1112 | Nitroglycerine | 55-63-0 | C(C(CON+[O-])ON+[O-])ON+[O-] | 2.5 | 227 | 13.3 | 2.05 |
| X2 | Oxybutynine | 5633-20-5 | O=C(OCC#CCN(CC)CC)C(O)(c1ccccc1)C2CCCCC2 | 3.9 | 357 | 57 | |
| 20332 | Scopolamine | 51-34-3 | CN1C2CC(CC1C3C2O3)OC(=O)C(CO)C4=CC=CC=C4 | 0.167 | 303 | 59 | 1.24 |
| X3 | Tulobuterol | 41570-61-0 | Clc1ccccc1C(O)CNC(C)(C)C | 0.5 | 228 | 93 | 0.82 |
| X4 | Selegiline | 2079-54-1 | C#CCN(C@@HC)C | 6 | 187 | <25 |
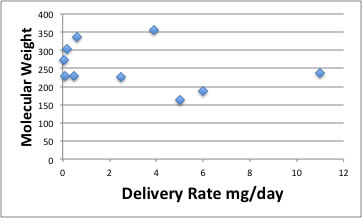
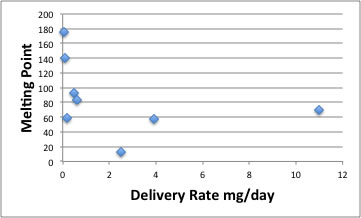
こうした薬が経皮でどれだけ吸収されるかは、「融点が低く、分子量が小さいほど、また適度に脂溶性を示す薬物が皮膚から吸収されやすいこと」が、「このテーブルから明らかであろう」と論文に記載してある。
上のテーブルをコピペして、グラフ化してみよう。
横軸のDelivery rateは個人差があるためか、範囲で示してあったが、最小量の値を採用した。
分子量の効果は下図のようになる。
大まかには分子量が小さい方が経皮吸収が大きくなるが、大きく外れるものもある。
融点の効果、
オクタノール/水分配比率(logKow)
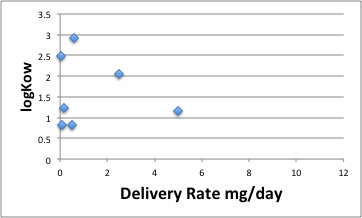
と思ったほどの相関は無い事がわかる。
これではある構造の薬を作った時にどのくらい経皮吸収されるか、構造のみから予測する事は困難だろう。
ハンセンの溶解度パラメータ(HSP)を使った検討
それでは、HSPを使って検討してみよう。
Hcode(ハンセンコード)やCAS#がある場合にはHSPiPソフトのデータベースを検索するか、DBに無ければ、薬のSmilesを用意してYMBで計算する。
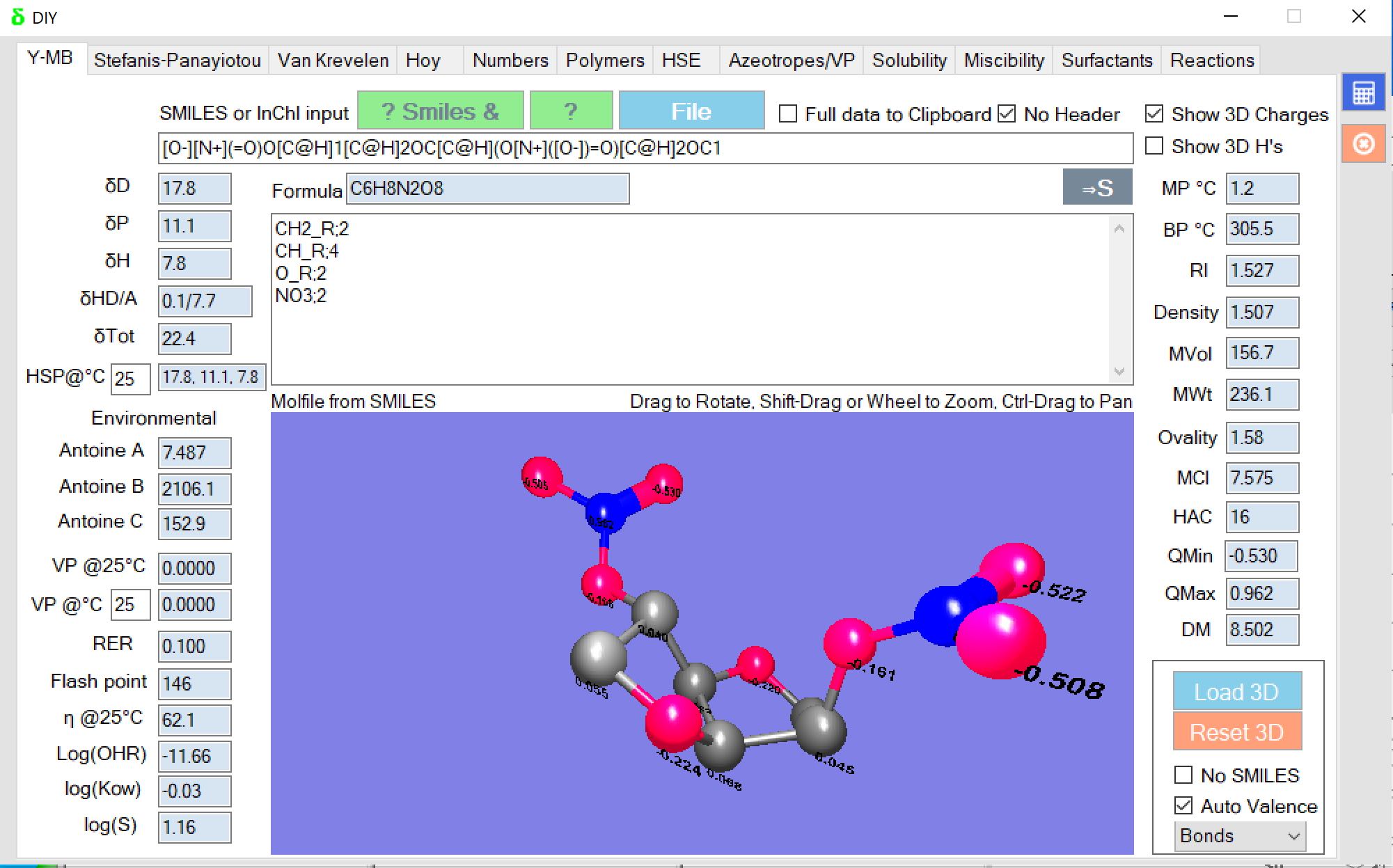
画面はHSPiP ver.5.2のYMBを使って、Smilesの構造式から、各種物性値を一括で計算した所だ。
HSPiPには、Smiles構造式の一覧を一括で計算する機能が搭載されている。
先のテーブルをタイトルごとコピーし、タブ区切りのテキスト・ファイル(SmilesTest.txt)としてセーブする。
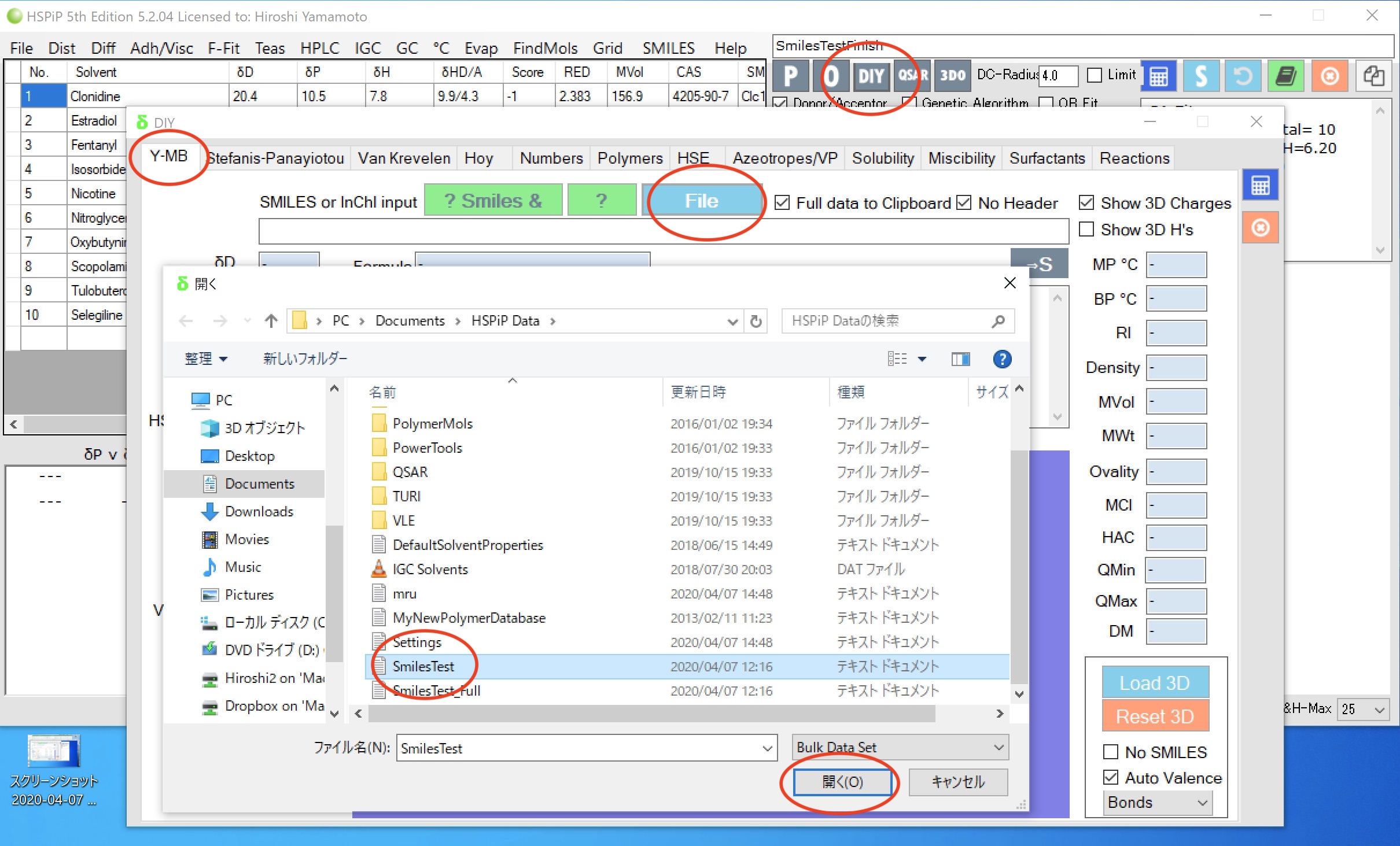
メイン画面から、DIY/YMB/Fileと辿り、先ほど作った、SmilesTest.txtを開くと自動的に解析が始まる。
そして、
SmilesTest.sofx
SmilesTest.hsdx
SmilesTest_Full.txt
の3つのファイルが作成される。
SmilesTest_Full.txtには、YMBで計算された全ての物性値が格納されている。拡張子、sofはSolvent Optimizer Fileという溶媒最適化に使うフォーマットだ。hsd拡張子はHansen Sphereを計算する用のフォーマットだ。
hsdxフォーマット
ここでは、メインプログラムからSmilesTest.hsdx を読み込んでみよう。
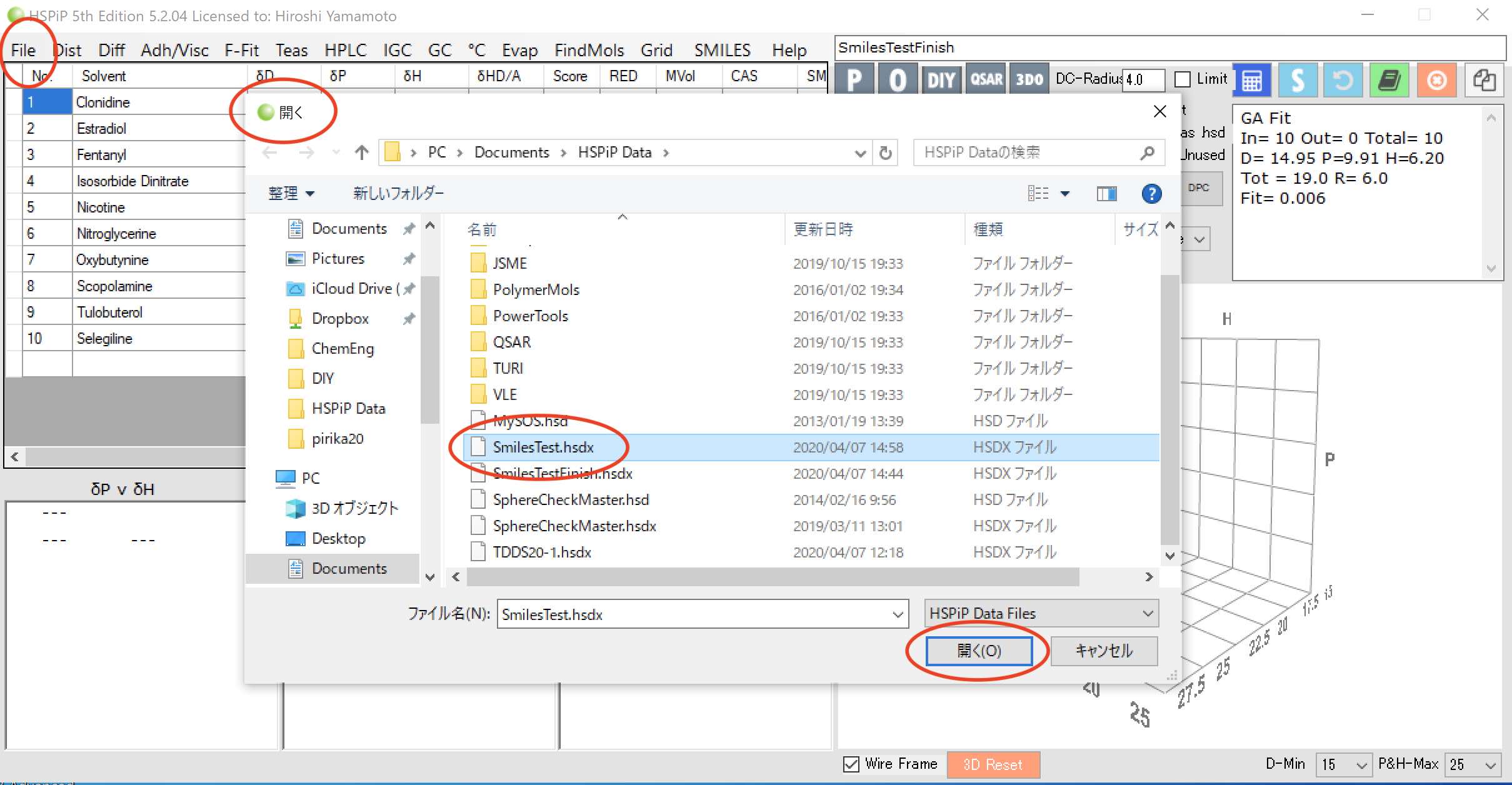
メイン画面からFile/開く/ファイル名を選択して読み込む。
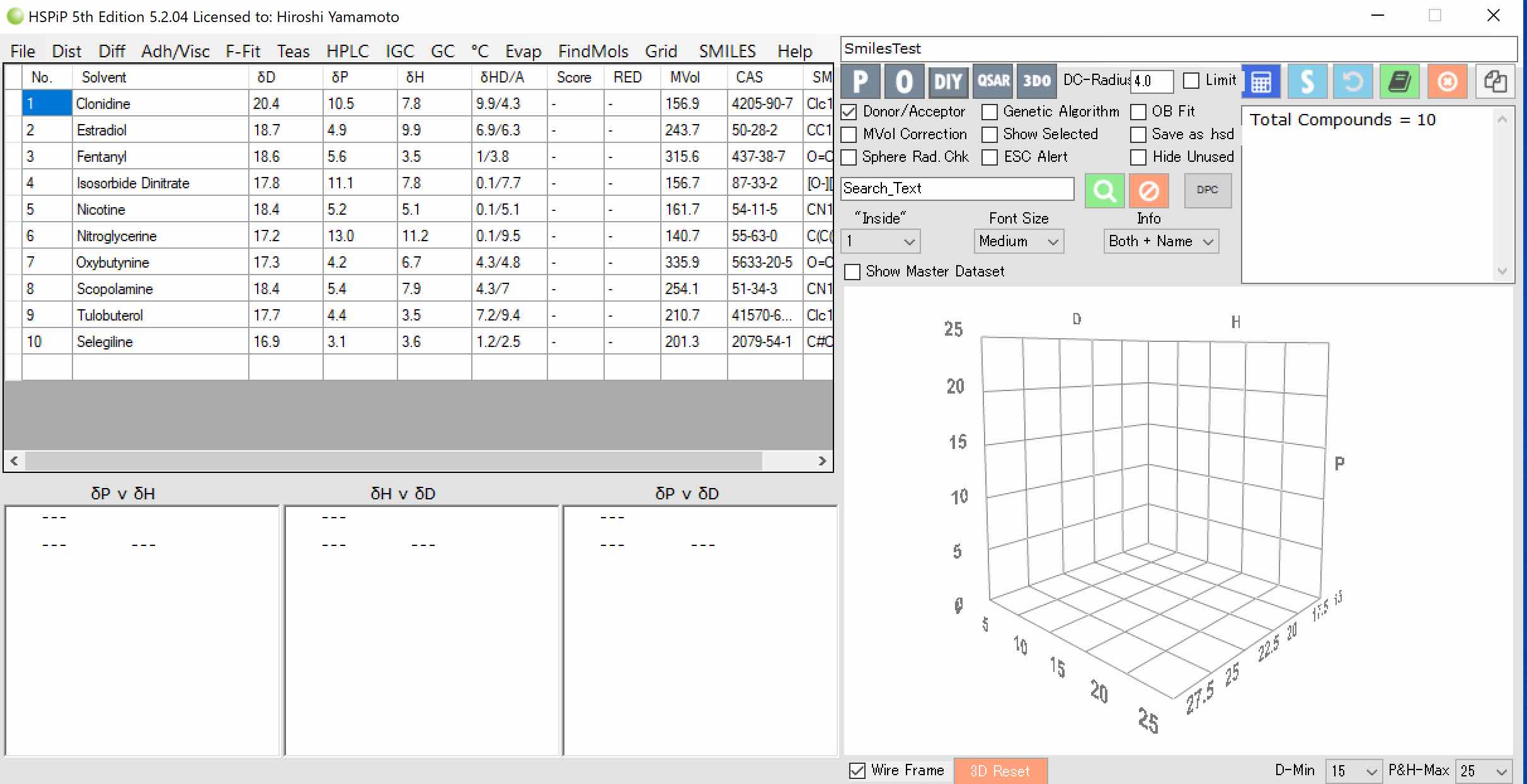
ここまで、準備ができたら、後はScoreを設定する。
HSPiPでは3種類のScoreを設定できる。
- 良溶媒に1, 貧溶媒に0を入れる。
- 1-6のScoreを入力して、良溶媒(1)と貧溶媒(6)の境のScoreを指定する。
- 実溶解度(g/100ccなど)をScoreにする。
ここでは単純に、Delivery rate mg/dayが1以上のものをScore=1,
それ以下のものをScore=0に設定する。
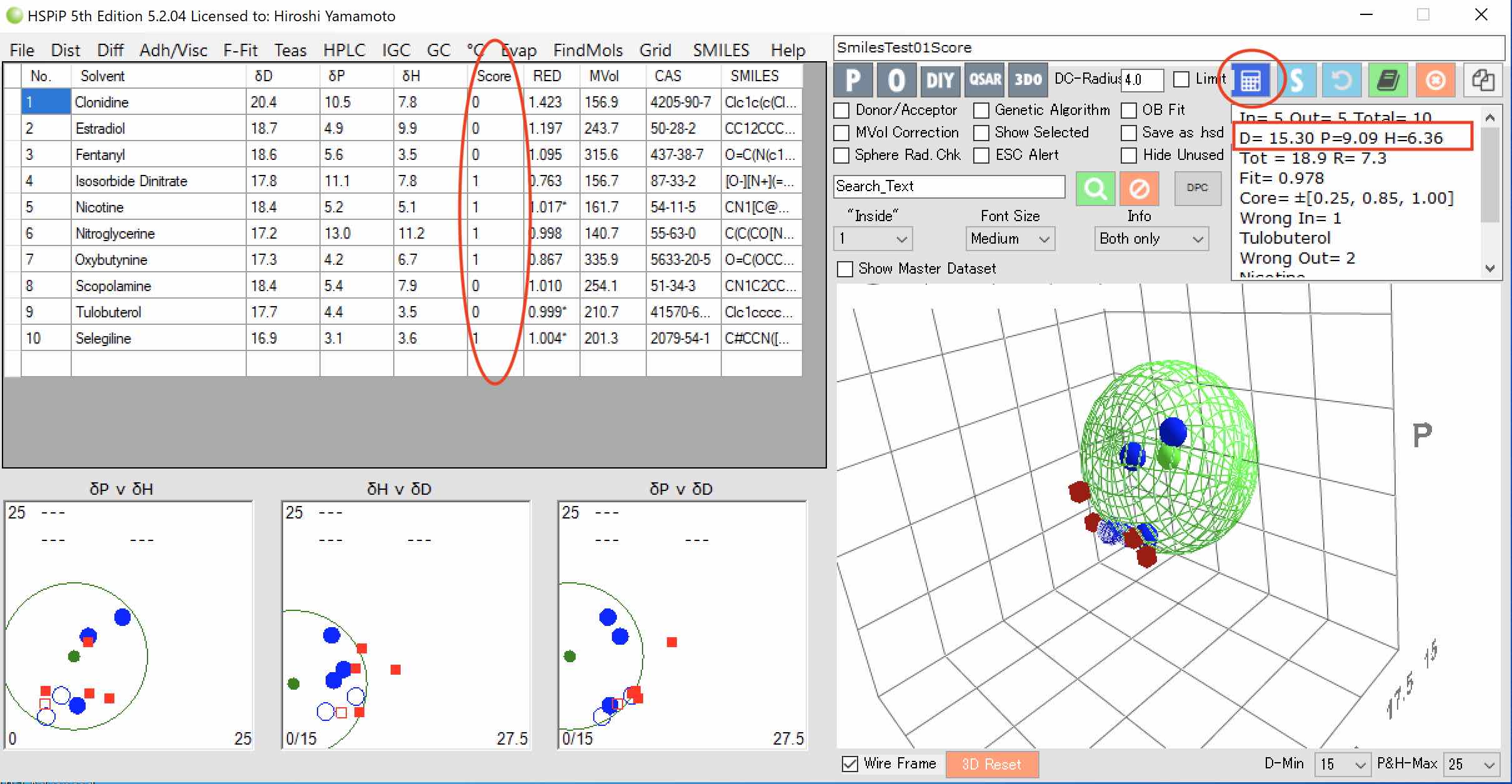
そしてSphereの計算ボタンをクリックすると、Score=1が球の内側、Score=0が球の外側になるような緑色のメッシュで示した「ハンセンの溶解球」を探索し、結果が表示される。
皮膚のHSPを[15.3, 9.09, 6.36]とした時の、皮膚から薬剤へのHSP距離は次式で計算することができる。
HSP距離
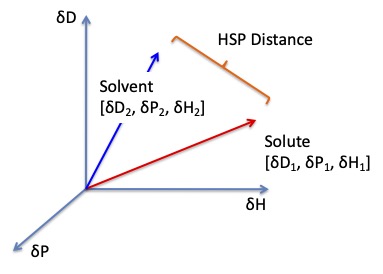
HSP distance(Ra)={4*(dD1-dD2)^2 + (dP1-dP2)^2 +(dH1-dH2)^2 }^0.5
(dDの前には4と言う係数が入ることに注意しょう。)
このHSP距離とlog(Deliv. rate)をプロットすると以下のようになる。
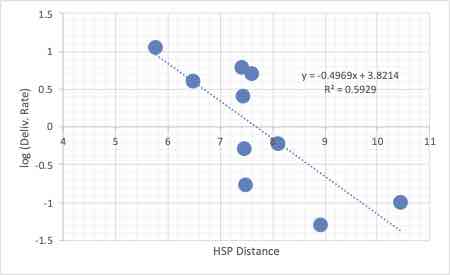
HSPの理論では、「HSPベクトルが近いほどよく溶解する」と言える。
図からはHSP距離が短いほど、log(Deliv. rate)が大きい右下がりの傾向がある事が確認できる。
しかし、HSP距離が大体7.5でありながら、log(Deliv. rate)が大きく変わる薬剤が5つほどあり、これらが相関係数を低くしている。
その理由は簡単で、分子の大きさが考慮されていないからだ。
最も簡単にHSP距離と分子の大きさの両方を取り込んだ式を評価する方法は重回帰式を作ることだ。
Scheme1= -0.520687*HSP距離 -0.003518*分子体積+4.770918
とすると、HSP距離と分子体積両方を加味した予測値を得る事ができる。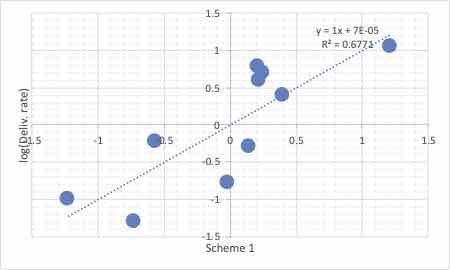
この精度が十分かどうかは、ひとまず置いておいて先に進む。
大事な事は、ここで示したリード化合物に対して、例えば水酸基をメチルエーテルに変えた時に、log(Deliv. rate)がどう変化するかを予測できるという事だ。
修飾した薬のSmilesの構造式さえあれば、YMBを使い特性値を計算、Scheme1で評価するだけだ。
それをコンピュータの中で高速に回して、候補化合物を創生し、良いもの順にソートして分子設計するのが、最近流行のマテリアルズ ・インフォマティクス (MI)という方法だ。
それを行うにはGUI(グラフィカル・ユーザー・インターフェイス)などはいらないので、CLIライセンスを導入して解析が行われている。
Delivery rate ではなく薬理活性でもやり方は同じだ。
では、このScheme1が得られたら、研究者は本当に安心して、論文や特許を書けるだろうか?
実際には、そんな論文や特許も多数存在する。
だが、たかだか、10個の実験値、決定係数 R^2 =0.67 程度ではかなり不安が残る。
そこで、HSPiPユーザーには更に高度な使い方を勧めている。
HSPiP のQSARツール
HSPiPのver.5からQSARツールが付属している。私が作ったツールだ。
表計算ソフトから、下図のように化合物の名称、Smilesの構造式、log(Deliv.rate)をクリップボードにコピーする。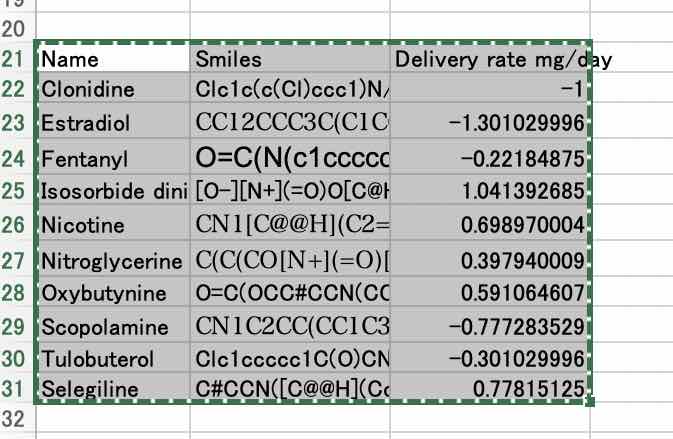
そして、メインプログラムからQSARボタンをクリックする。
そしてクリップボードからペーストボタンをクリックすると先ほど選択したテーブルがQSARパネルにペーストされる。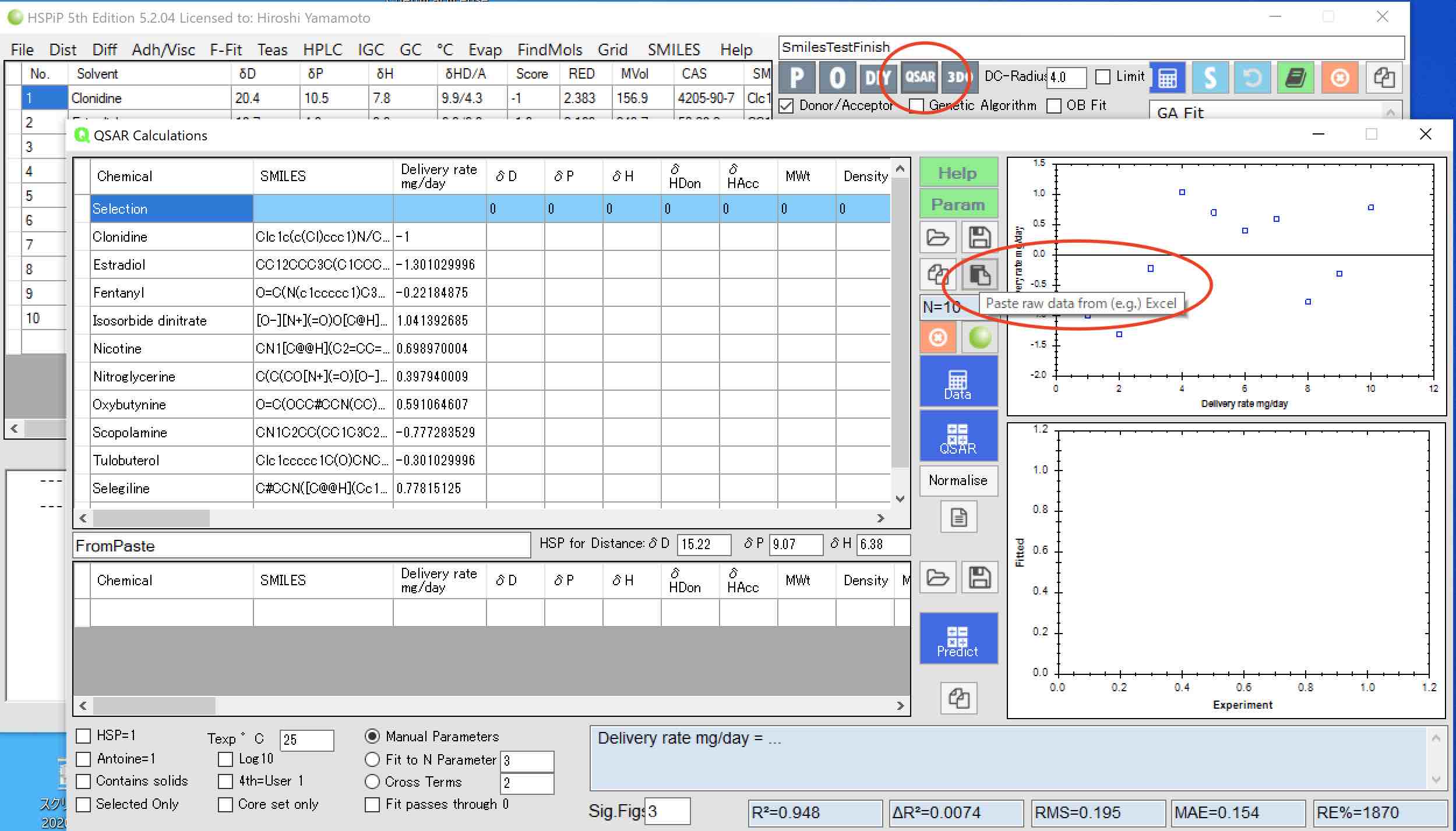
Dataボタンをクリックすると、Smilesの構造式から様々な物性値を推算する。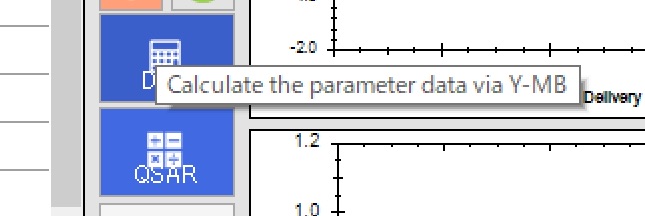
推算された物性のタイトルの部分をクリックすると、3列目(Deriv.Rate)とのグラフが表示される。どのような物性値と単相関があるのかを簡単にチェックする事ができる。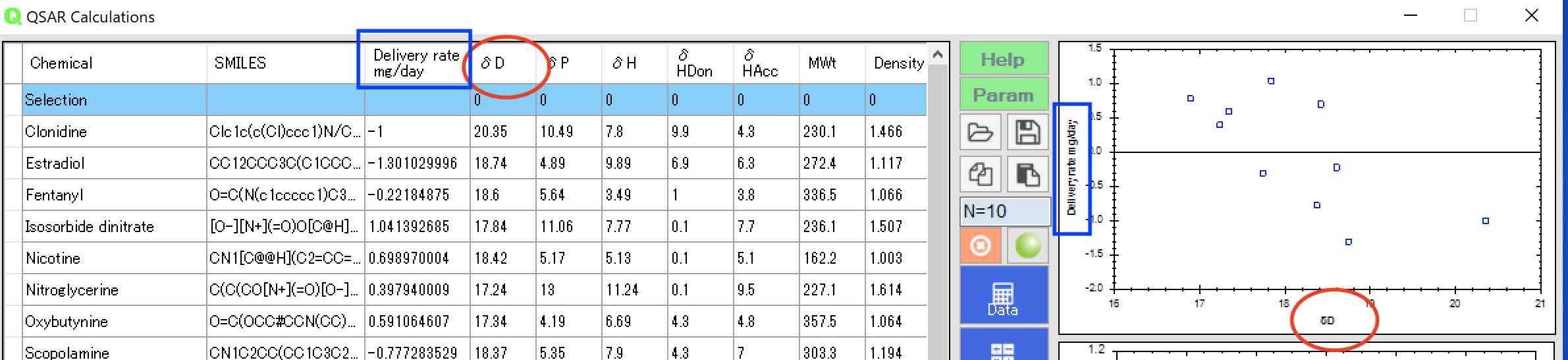
準備ができたら、HSPiPにQSARをやらせる。
一番単純には、Fit to N Parameter に3とか4を指定してQSARボタンをクリックする。
すると、YMBが作り出した様々な物性値から3変数を選択して予測式を構築してくれる。
どの変数を選択するかは、単純に相関係数が高くなるように選んでいるだけだ。
これをベースに削ったり、付け加えたりしながらより合理的な推算式を構築していく。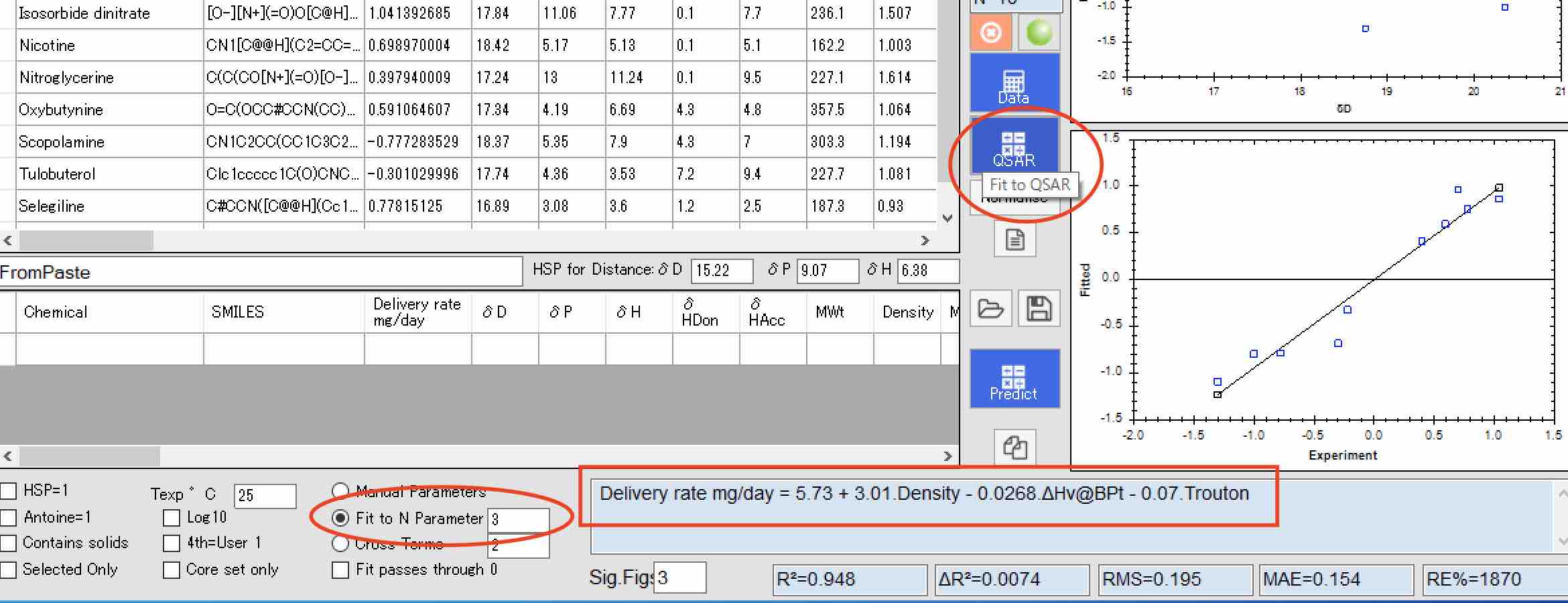
ここで3変数を自動選択させた場合は、次のようなScheme2が作成された。
Scheme2 = 5.73 + 3.01.Density – 0.0268.ΔHv@BPt – 0.07.Trouton
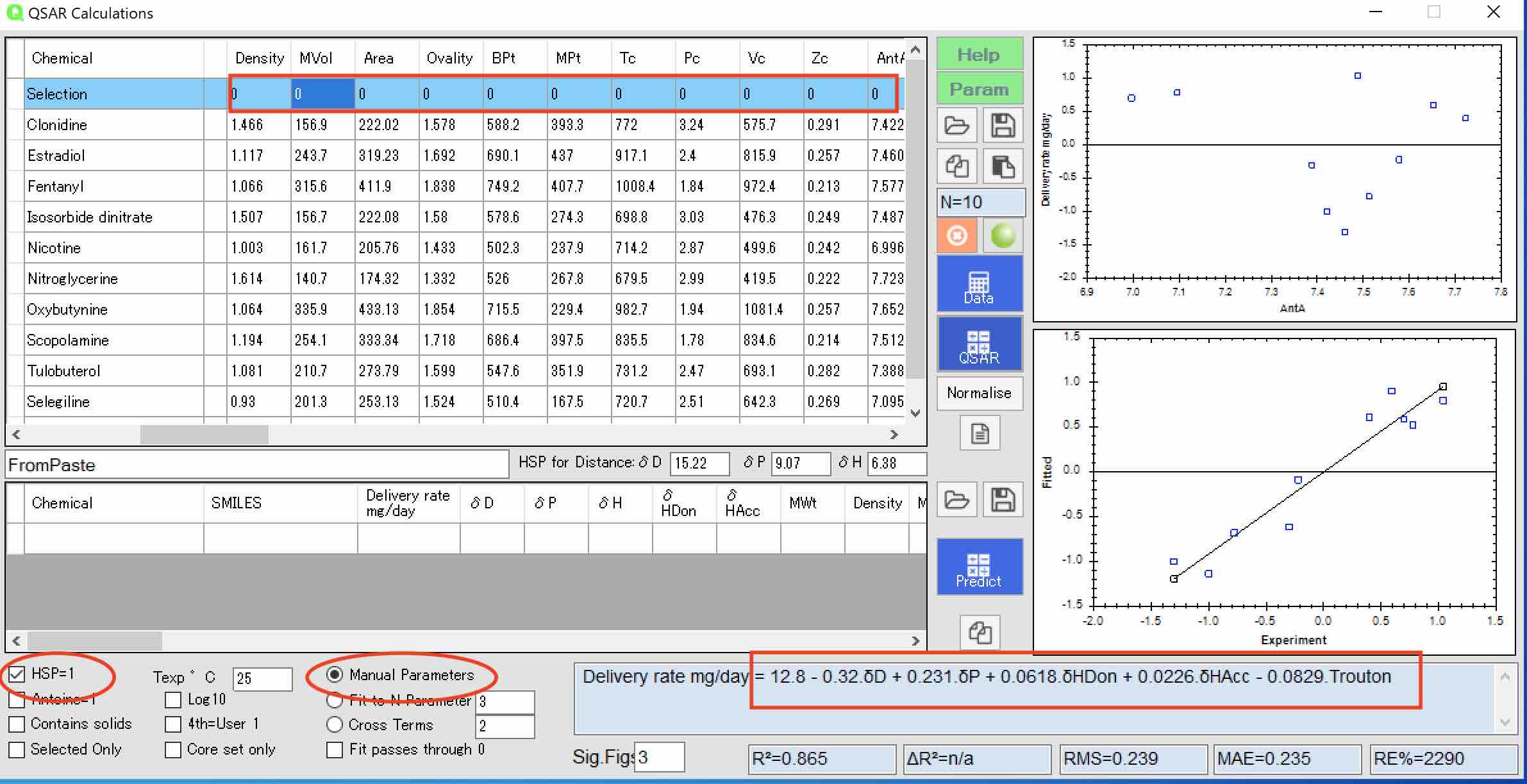
HSPの値を使い、他の値はマニュアルで選択してみる。
水色の領域をダブルクリックすると、値が0,1で切り替わる。1の時にはその物性値を使ってQSAR式を作る。
Scheme3 = 12.8 – 0.32.δD + 0.231.δP + 0.0618.δHDon + 0.0226.δHAcc – 0.0829.Trouton
このようにHSPiPのQSAR機能を使って様々な種類のQSAR式を作成する。
そして、それらの「ある程度相関係数が高い」式で常に予測値が高くなる構造は、高い確率でDeriv.Rateが高くなると言える。
データが少ない分、予測性能を上げようとする場合には、このような工夫が必要になる。
臨界定数などをベース。
Antoine定数などをベース
溶解性などをベース
熱力学物性値などをベース
慣れてくると、いろいろな式が自由に作れるようになってくる。
これは、自動化されたAIに人間が対抗できる大事なやり方だと思う。
pirika.comで化学 > 化学全般
> 次世代HSP2, YMB Pro for MI >
> 解析例トップ
DIYトップページ >
医薬品・化粧品トップページ > 経皮吸収型ドラッグデリバリーシステム
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください。
