2022.10.10改訂(2012.4.25)
pirika.comで化学 > 化学全般
> 次世代HSP2, YMB Pro for MI >
> 解析例トップ
DIYトップページ
医薬品・化粧品トップページ > ヒスタミン受容体拮抗剤
HSPiPのユーザーからおもしろい提案を受けた。
辛いもののレセプター、匂いのレセプター、昆虫のレセプターがHSPと関係があるのなら、ヒスタミンのレセプターもHSPに関係があるのでは無いかというものだ。
化学増刊No.116の創薬のための分子設計という本のN-カルバモイルアミジン誘導体のH2-遮断活性のテーブルを送って下さった。
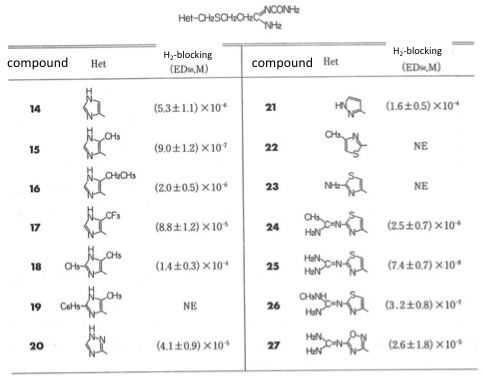
右の部分は同じなので、左の部分(Het)の違いによってH2-遮断活性が異なってくる。
テレビなんかで宣伝している”先回りしてブロック”をイメージすればいいのだろうか?
先回りするには、Hetの部分が受容体にどれだけ溶けやすいか?が問題になるかもしれない。
そこでハンセンの溶解度パラメータ(HSP)が”似たものは似たものを溶かす”の原理が有効に使えないか?という話しらしい。
そこでHetの部分のHSPを計算してみる。
そのためには、各Het構造のSMILESが必要になる。
データベースには入っていないのでSMILESは自分で作らなくてはならない。
JSMEと言うお絵かきソフトがpirikaのwebページにも置いてあるから試しに使ってみるのも良いだろう。
データはコピペできるように提供するが、自分でも組めるようにしておこう。
無精をすれば、それは自分に跳ね返る。
テーブル
| No | Smiles | H2Block | MinusLOG(H2B) |
|---|---|---|---|
| 14 | C1=NC(=CN1)C | 5.30E-06 | 5.27572413 |
| 15 | CC(N=C1)=C(N1)C | 9.00E-07 | 6.045757491 |
| 16 | CC(N=C1)=C(N1)CC | 2.00E-06 | 5.698970004 |
| 17 | FC(C(N1)=C(N=C1)C)(F)F | 8.80E-05 | 4.055517328 |
| 18 | CC(N1)=C(C)N=C1C | 1.40E-04 | 3.853871964 |
| 19 | CC(N1)=C(C)N=C1C2=CC=CC=C2 | ||
| 20 | C1=NC(=NN1)C | 4.10E-05 | 4.387216143 |
| 21 | N1N=C(C=C1)C | 1.60E-04 | 3.795880017 |
| 22 | CC1=CSC(=N1)C | ||
| 23 | CC(CS1)N=C1N | ||
| 24 | NC(=NC1=NC(=CS1)C)C | 2.50E-06 | 5.602059991 |
| 25 | NC(=NC1=NC(=CS1)C)N | 7.40E-08 | 7.13076828 |
| 26 | NC(=NC1=NC(=CS1)C)NC | 3.20E-07 | 6.494850022 |
| 27 | NC(=NC1=NC(=NO1)C)N | 2.60E-05 | 4.585026652 |
活性はlogを取って符号を逆転してある。(活性の高いものは少ない量で効くための補正)
そして定量的なSphereを求める。
Sphere(球)というのはHansen先生が導いた概念で、ある溶質をよく溶解する溶媒のHSPベクトルを3次元空間にプロットすると、よく溶かすものはHSPベクトルが似ていて、3次元空間(ハンセン空間)で集まってきて球を構成する。
未試験の溶媒のHSPベクトルがその球の中に入ってくるなら、それもよく溶解するだろう、という概念だ。
その”良く”というのの概念が球の半径を規定する。
そこでHetの部分についてHSPベクトルを計算し、活性が高いものから、低いものまでHSP距離と相関が高くなるように球の中心を定めてあげる。(定量的なSphereのページを参照のこと)
2022.10.9 大失敗。
ページを公開してから10年。
大間違いが発見された。
これだから、査読もないWebページなんて価値ないと言われそうだ。
ここでは、
活性はlogを取って符号を逆転してある。(活性の高いものは少ない量で効くための補正)
をしているのを忘れていた。
お詫びに、すぐに計算できるように、hsdxフォーマットのデータを提供しよう。
Histamine.hsdxとセーブしてHSPiPから開いてみよう。
これを、HSPiP 5.4から開くと次のようなる。
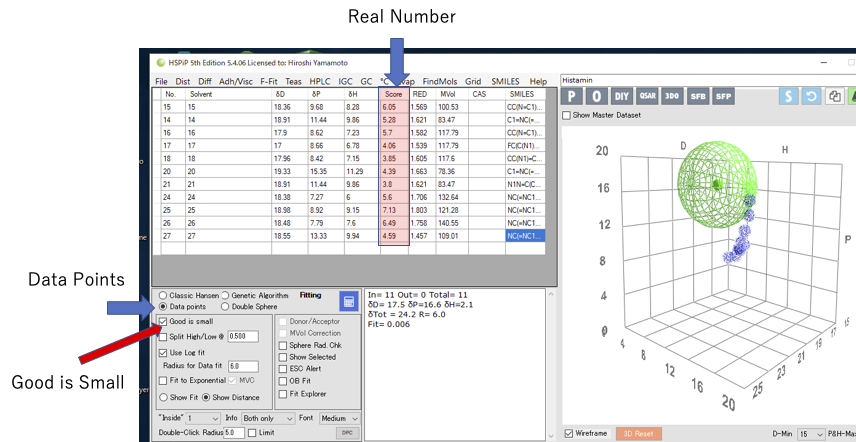
Scoreの部分には、遮断活性の実数が入っている。
そこでSphereの計算は、Data Pointsにしなければいけない。
そして、大事なポイント、この実数は、活性が高いほど値が小さくなる。
(少量で高い活性になる)
そこで、Good is Small にチェックを入れなくてはいけない。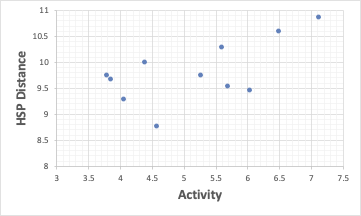
そのようにして得られたSphereの中心からの距離とActivityをプロットすると上図のようになる。
活性が高い(値が小さい)ほど、HSP距離が短くなる。
HSP距離
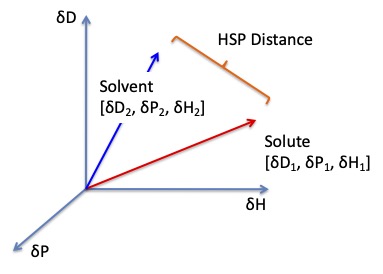
HSP distance(Ra)={4*(dD1-dD2)^2 + (dP1-dP2)^2 +(dH1-dH2)^2 }^0.5
(dDの前には4と言う係数が入ることに注意しよう。)
いくつか大きく乖離しているものがある。
本来、この系ではδHacid, δHbaseを導入しなくてはならないのだと思う。
Drag=回転, Drag+Shift キー=拡大、縮小, Drag+コマンドキーかAltキー=移動。小さな球をクリックすればHetの番号が現れる。
ここで大きな緑の球はレセプターを示している。
この球の中心がレセプターのHSP[17.5, 16.6, 2.1]と考えられる。
ただし、この系は、Electron Donor/Acceptorを導入して考えなくてはダメなようだ。
次期改訂ではそこまで踏み込むつもりだ。
間違い記述
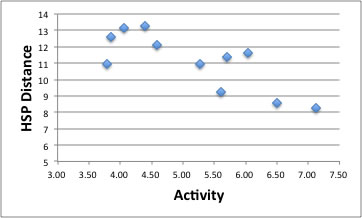
すると求まった結果は上のグラフのようになる。(HSPiPを持っている場合には最新のパラメータで計算し直してみよう。)
球の中心がレセプターのHSPになる。かなり高い相関で、HSP距離の短いもの(よく溶けやすいもの)は高活性であることがわかる。
それではHSP以外ではどんな項目が活性に影響を与えているのだろうか。例えば下のグラフのように分子体積も相関がある。
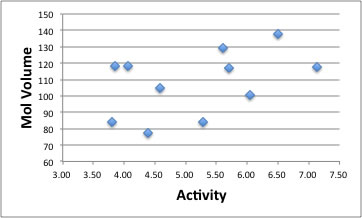
そこで、大学での講義に使った変数選択重回帰法を使って、変数を選択してQSARモデルを作成してみる。
一番重要なのは溶解性を表すHSP距離だが、分子体積、logBCF(生物濃縮性)、表面張力であった。
その4変数を用いて重回帰式を構築すると下のように結構な精度で活性を予測できる。
(logBCFについてはpirikaのこちらの記事を参照。
表面張力についてはこちらの記事を参照)
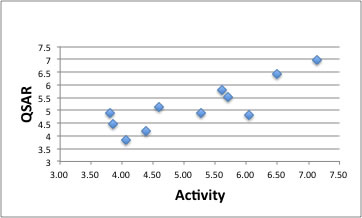
活性が低い所で、相関が低いが、低い所はそもそも欲しい領域では無いので、大きな問題では無い。
それでは、HSPiPのデータベースの中にある化合物でこのレセプターに近いHSPを持つものはどんなものがあるだろうか?検索してみた。
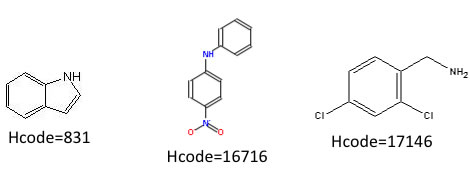
分子体積、logBCF(生物濃縮性)、表面張力は別途計算しなくてはならないが、リード化合物としては面白いかもしれない。
日本での医薬品開発にはHSPはまだそれほど注目されないが、欧米では急速に利用が進んでいるのはこうしたことが簡単にできるからであろう。
自分は奥さんと子供がアレルギー体質なのでいい薬がどんどん開発されることを願ってやまない。
Drag=回転, Drag+Shift キー=拡大、縮小, Drag+コマンドキーかAltキー=移動。小さな球をクリックすればHetの番号が現れる。
ここで大きな球はレセプターを示している。Hetが球で表されているものは溶解度が大きなもの。立方体で表されているものは溶解度が小さなもの。
色はdHdoとdHacを表している。赤色はdHdo性が高いもの。青色はdHacが高いもの。色の強さはdHの絶対値を示している。
この系は、本来、水素結合項はドナーとアクセプターに分割して考えたほうが良いと思う。
pirika.comで化学 > 化学全般
> 次世代HSP2, YMB Pro for MI >
> 解析例トップ
DIYトップページ
医薬品・化粧品トップページ > ヒスタミン受容体拮抗剤
Copyright pirika.com since 1999-
Mail: yamahiroXpirika.com (Xを@に置き換えてください)
メールの件名は[pirika]で始めてください
