隠遁Chemistと愛(AI)の交換日記
原子団寄与法(Group Contribution)法は原子団を分割した段階で結合情報が失われてしまう。「原子団寄与法を再定義する」でJobackの原子団をベースにした物性推算法を紹介した(JBDGC)。
結合情報が失われない為、物性推算の精度はとても高い。なんといっても、pKaのようにCOOHの先に何がついているかで値が変わる、そのような物性で大きな効果がある。
基本設計は2015年ごろ行ったので古い。
今回Pirika Proに実装するようにいくつかの原子団の拡張を行った。
LLC: Pirika.com社CEOの山本博志の基本的なポリシーは、DGCを実際に使ってみる。合わないものをなぜ合わないのか考える。どう改良したら合うようになるか試す。
それを繰り返す。それが大事だ。
DGC法の一番のポイントは、例えば分子中にCH3があったらCH3がどんな原子団と結合しているかによってCH3の数え方を変える点だ。
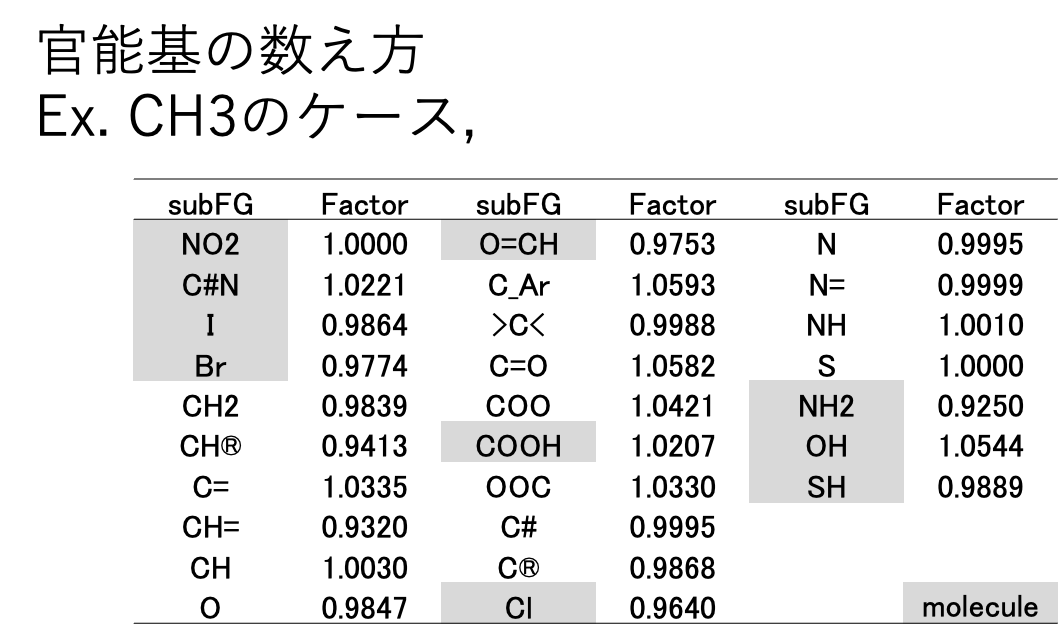
JBDGCでは、メインの59原子団がどんな原子団と接続しているかを調べる。相手によって原子団の数え方を変える。相手によっては分子そのものになる。
CH3NH2の場合、CH3は0.9250個と数える。
結合相手、係数は解析したDataSetに依存する(Dynamicに変化してしまう)。
非常に精度は高くなるが、例えばハロゲン原子で問題が出る。
例えばCH3-CF3という分子を考える。CF3という原子団は定義されていない。
そこで、CH3は4級炭素と結合していると数えるしかない。
>C<の係数は0.9988になる。>C<に結合するCH3は0.9988と数える。
CF3でもtBuでも>C<に変わりはないのでCH3の数え方は同じになる。
それで問題がでる物性値もある。
CF3COOHのような化合物のpKaで問題が出ていた。
COOHが>C<に接続したと考えるとCOOHがCF3によって電子が引っ張られるという効果は導入されない。>C<_F_F_Fの時に>C<の数え方が大きく変わるかが問題になる。
もともと、pKaに影響を与えるのは活性水素を持つような原子団だけだ。それ以外はほとんど影響を与えない。たとえば、CH3CH2CH2CH3とかCF3CF2CF3はpKaに何の影響も与えない。そこで>C<_F_F_Fがあっても>C<の数え方はあまり大きくは変わらない。
説明は難しいが、何を言いたいかというと、JBDGCの原子団を拡張する時の手間を削減したい。
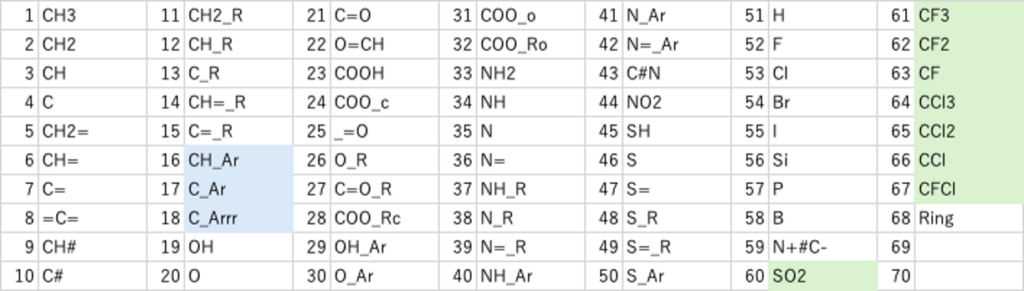
今回、SO2, CF3, CF2, CF, CCl3, CCl2, CCl, CFClの原子団を増やした。
SMILESの構造式から原子団を数え上げてテーブル化する。
原子団同士の結合情報をもとにDGCで推算式を構築する。
推算式を用いて任意の化合物の物性を推算する。
原子団の定義の増減によってプログラムの修正が必要ないようにプログラムの修正を行った。
ユーザーが持つデータをDGCで推算式を作る。原子団の拡張が伴う事がある。
たとえばN+やCl-のようなDES用の拡張が簡単にできるようにする。
準備が整った。
多分YMBの物性推算式は全面的に置き換えることになるだろう。
ダイナミックな変更が簡単にできる。楽しみになった。
